Última actualización: 17 de julio de 2021
El genoma del SARS-CoV-2 está formado por un ARN de casi 30.000 nucleótidos. Cada uno de esos nucleótidos puede contener una molécula de lo que se llama base nitrogenada, habiendo cuatro distintas: Adenina (A), Uracilo (U), Citosina (C) y Guanina (G).
Ese ARN contiene las instrucciones necesarias para formar sus proteínas, que son una secuencia de unos compuestos denominados aminoácidos. Estos aminoácidos

El ARN contiene por así decirlo para poder infectar células humanas y replicarse. Los virus con ARN tienden a presentar múltiples mutaciones conforme se multiplican (es decir, la secuencia de nucleótidos del ARN no se copia exactamente igual, sino que hay algún cambio), sin corregir los errores que hay durante la replicación del ARN en la multiplicación, lo que puede llevar a cambios impredecibles en el comportamiento del virus, incluyendo su letalidad o su tasa de contagio. Sin embargo, los coronavirus suelen ser una excepción, y suelen ser más estables, aunque sus mutaciones pueden ir acumulándose.
Una mutación en el ARN del virus (cambio de una base nitrogenada, pérdida de nucleótidos, etc) puede provocar (no siempre) que haya cambio en la secuencia de aminoácidos de las proteínas que contiene, pues se sintetizan a partir de la información del ARN, y este cambio en las proteínas puede afectar a la función de las mismas, y por tanto, al funcionamiento del virus. Las mutaciones pueden desaparecer o propagarse. Algunas mutaciones no tienen mayor importancia, pero otras sí que la pueden tener, en la medida en la que la función de las proteínas se vea alterada.
El genoma (ARN) del virus por tanto contiene instrucciones para fabricar sus proteínas. Aproximadamente dos terceras partes codifican las proteinas ORF1a y ORF1b. Con respecto a la proteína espiga S, la que usa el virus para entrar en nuestras células, parece que se "fabrica" a partir de una secuencia de 4.000 nucleótidos del ARN del virus. Por tanto, un cambio en esa secuencia del ARN podría cambiar la proteína espiga S, y la forma de que ésta pueda entrar a las células de nuestro organismo. Algunos de estos cambios podrían potencialmente afectar a la infecciosidad, a la resistencia del virus a medicamentos o vacunas, etc, aunque parece poco probable, pues el mecanismo que tiene en la actualidad ya le permite entrar en las células de nuestro organismo. El resto está orientado a codificar las proteínas E, M, N y otras proteínas accesorias.

Fuente: The New York Times
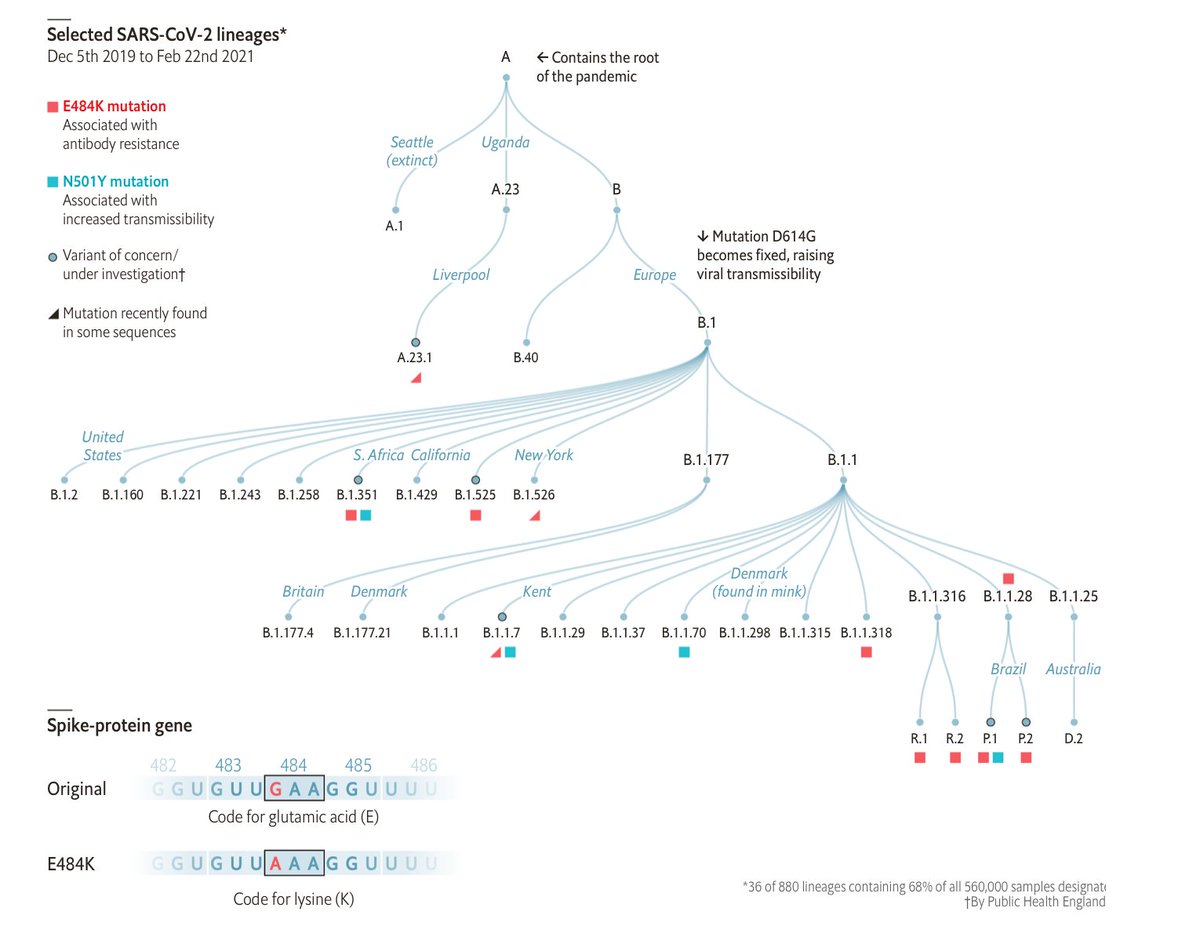
A medida que ha ido avanzando la pandemia, se han ido secuenciando virus, de forma que se han ido documentando las mutaciones a través de un linaje, que es una rama del árbol genealógico viral. Un grupo de coronavirus que comparten el mismo conjunto heredado de mutaciones distintivas se denomina variante. Si se acumulan suficientes mutaciones en un linaje, los virus pueden desarrollar diferencias claras en su funcionamiento. Estos linajes se conocen como cepas. Una cepa por tanto, estrictamente hablando, sería una variante con un fenotipo demostrablemente diferente, como por ejemplo, una diferencia en antigenicidad, transmisibilidad o virulencia. En el transcurso de la pandemia, han surgido varias variantes del SARS-CoV-2. Algunos de ellos están generando preocupación de que puedan prolongar la pandemia o hacer que las vacunas sean menos efectivas. Dentro de las variantes, se puede hacer distinción entre:
- Variantes normales
- Variante de interés (VOI): son variantes que pueden tener su interés ante un potencial comportamiento distinto, pero no demostrado. Si se demostrara, pasarían a ser variantes de preocupación.
- Variante en investigación (VUI): es el paso previo de una variante de interés a ser una variante de preocupación. Se estudia más en profundidad, y se decide si se eleva a la categoría de VOC.
- Variante de preocupación (VOC): son variantes que han aumentado la transmisión, la virulencia o pueden evadir la inmunidad (ya sea natural o derivada de una vacuna) y las medidas de salud pública, incluidos los diagnósticos y los medicamentos.
Variantes de preocupación |
||
|---|---|---|
| Linaje | Nombre de variante | Estado |
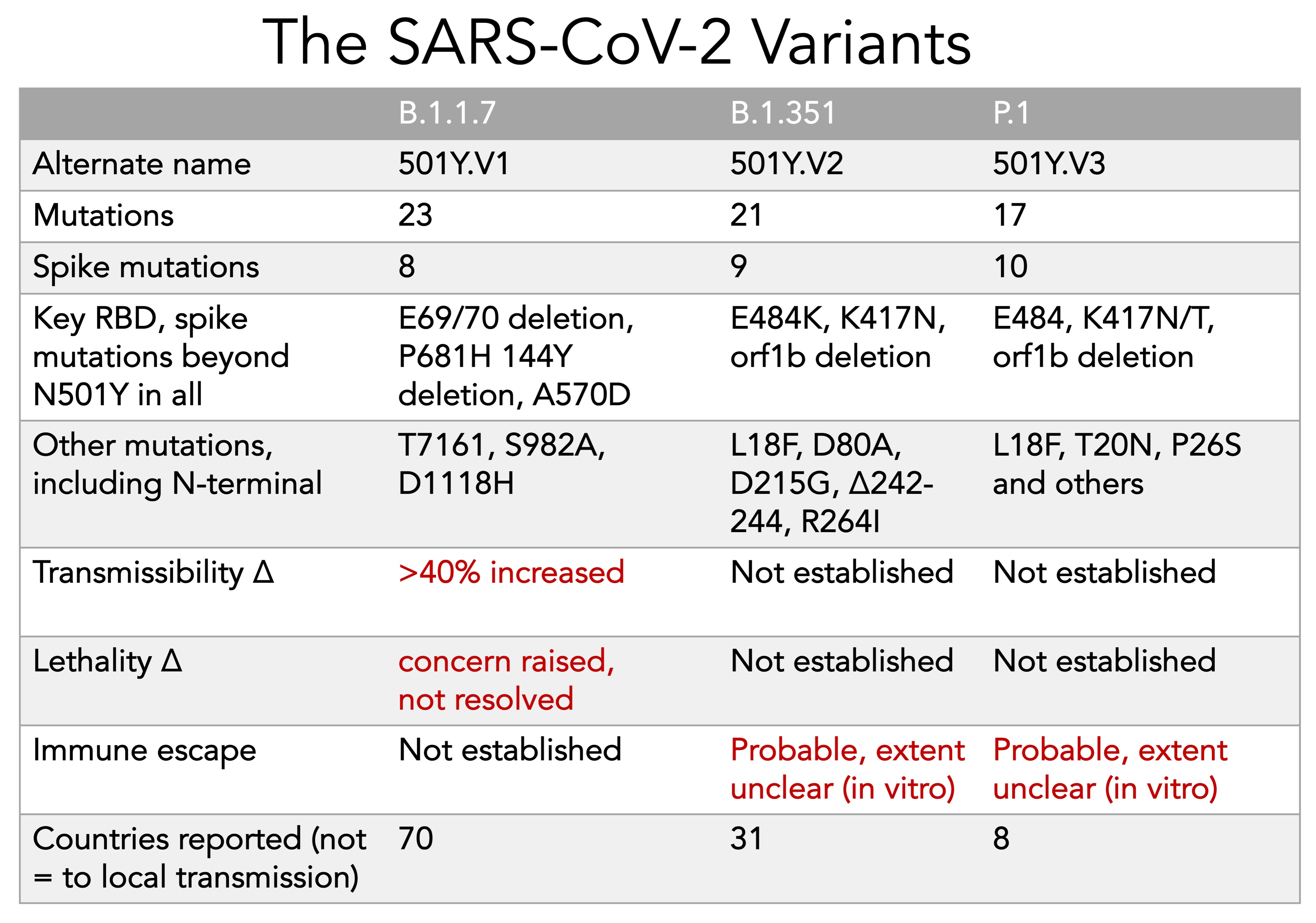
| B.1.1.7 | Variante de preocupación 202012/01 o 501Y.V1 | Surgió en Gran Bretaña en diciembre y es aproximadamente un 50 por ciento más infeccioso. Ahora se detecta en más de 70 países y 33 estados. |
| B.1.351 | 501Y.V2 | Surgió en Sudáfrica en diciembre. Reduce la eficacia de algunas vacunas. |
| P.1 | 501Y.V3 | Surgió en Brasil a finales de 2020. Tiene mutaciones similares a B.1.351. |
Mutaciones que pueden ayudar a que el coronavirus se propague |
||
| Linaje | Mutación | Estado |
| B.1 | D614G | Apareció a principios de 2020 y se extendió por todo el mundo. |
| Varios | N501Y | Una mutación definitoria en varios linajes, incluidos B.1.1.7, B.1.351 y P.1. Ayuda a que el virus se una más fuertemente a las células humanas. |
| Varios | E484K | Aparece en varios linajes. Puede ayudar al virus a evitar algunos tipos de anticuerpos. |
| Varios | L452R | Cada vez más común en California, pero aún no se ha demostrado que sea más infeccioso. |
Otras variantes en la noticia |
||
| Linaje | Nombre de variante | Estado |
| B.1.427, B.1.429 | CAL.20C | Lleva la mutación L452R . Común en California, pero aún no se ha demostrado que sea más infeccioso. |
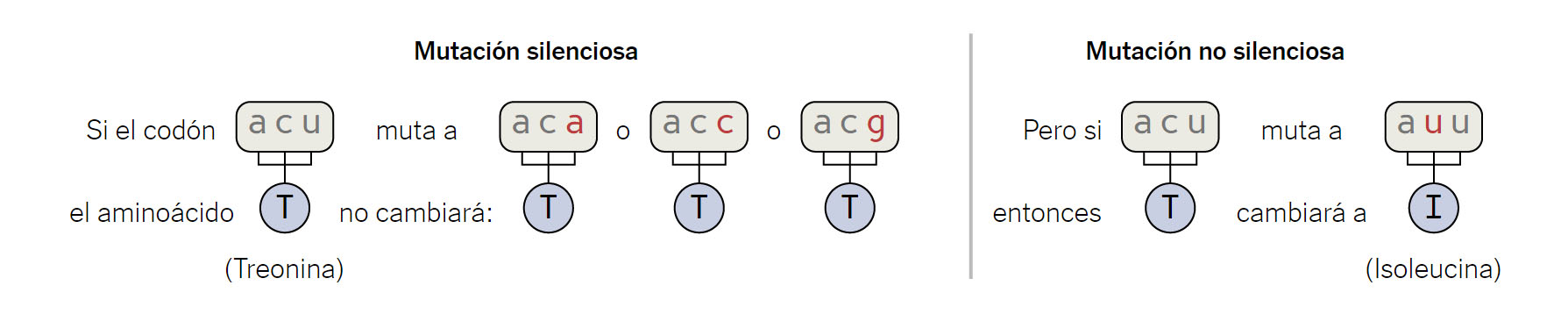
Las mutaciones se van documentando con dos letras y un número, que pueden hacer referencia a un cambio de bases nitrogenadas en el ARN o a un cambio de aminoácidos en la proteína espiga S; por ejemplo, una variante A100B significa que en la posición 100 de la proteína ha cambiado un aminoácido A por uno B. Así, en España se detectó en verano la variante A222V, donde la proteína espiga cambió en su posición 222 el aminoácido alanina (A) por valina (V). Puedes ver más sobre las mutaciones aquí. Esta variante se expandió por toda la geografía europea durante el verano (ver estudio). La primera mutación se documentó el 8 de enero de 2020. Con respecto al primer genoma secuenciado, se observó que en el ARN cambió una Citosina por un Uracilo en la posición 186, no provocando esta mutación ningún cambio en ninguna proteína (lo que se conoce como mutación silenciosa).

Fuente: The New York Times
El 27 de febrero se observó en otra secuencia dos mutaciones más en el ARN, además de la anterior, apareciendo nuevos uracilos. Estas mutaciones sí provocaron cambios en las proteínas (no en la proteína espiga S), apareciendo un cambio de aminoácidos (isoleucina I en proteína ORF1a y tirosina Y en proteína E) en dos posiciones donde había otros. Esto es lo que se conoce como mutaciones no silenciosas. Hay situaciones en las que un cambio de una única base nitrogenada no cambia el aminoácido que se va a generar, pues los aminoácidos se codifican a partir de una terna de bases nitrogenadas, y existen aminoácidos que se codifican con varias combinaciones distintas. Pero en otros casos sí que puede afectar el cambio de una base y producirse un cambio en un aminoácido de una proteína, por lo que estas mutaciones se llaman no silenciosas.

Fuente: The New York Times
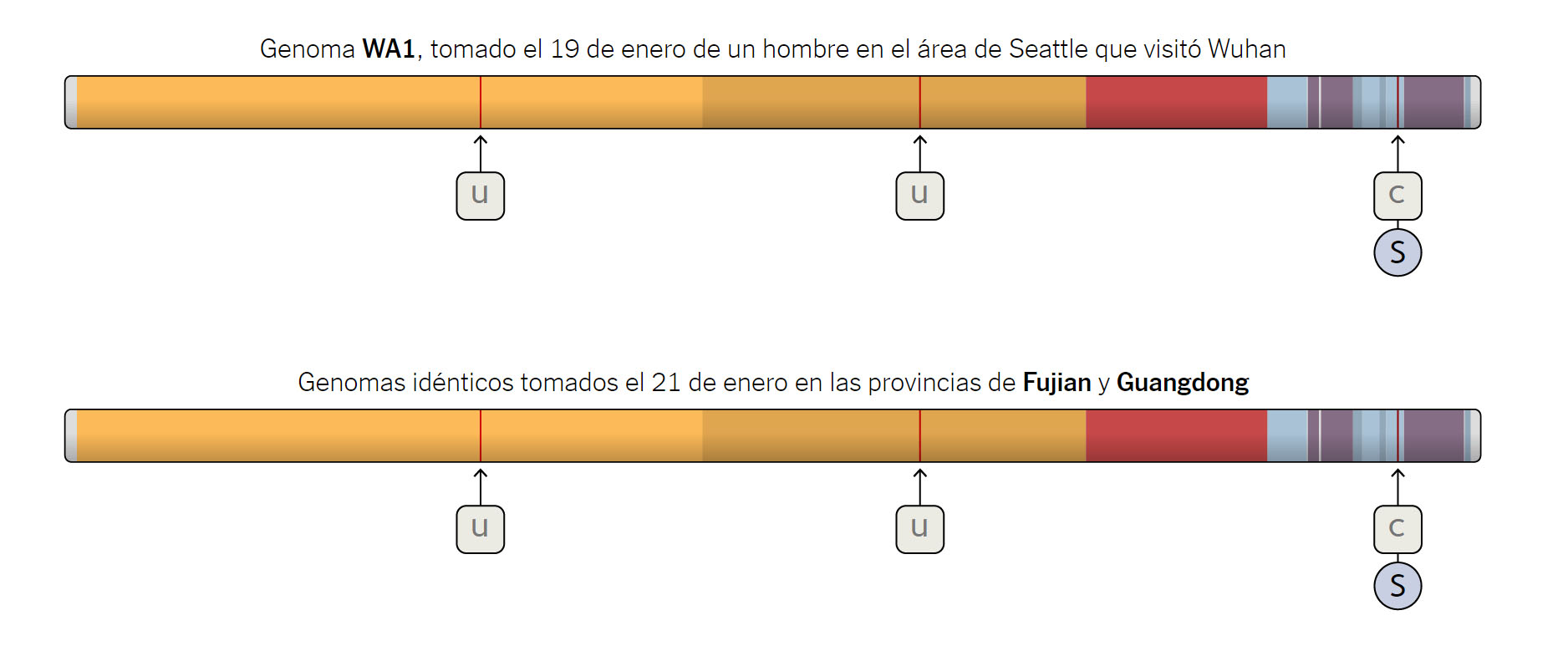
El estudio de estas mutaciones contribuye a saber cómo se propaga el virus. Por ejemplo, en días muy cercanos se secuenció el mismo genoma en Seattle (EEUU) y China. La persona que lo tenía en EEUU había vuelto de Wuhan, en lo que es un claro caso de contagio importado.
Fuente: The New York Times
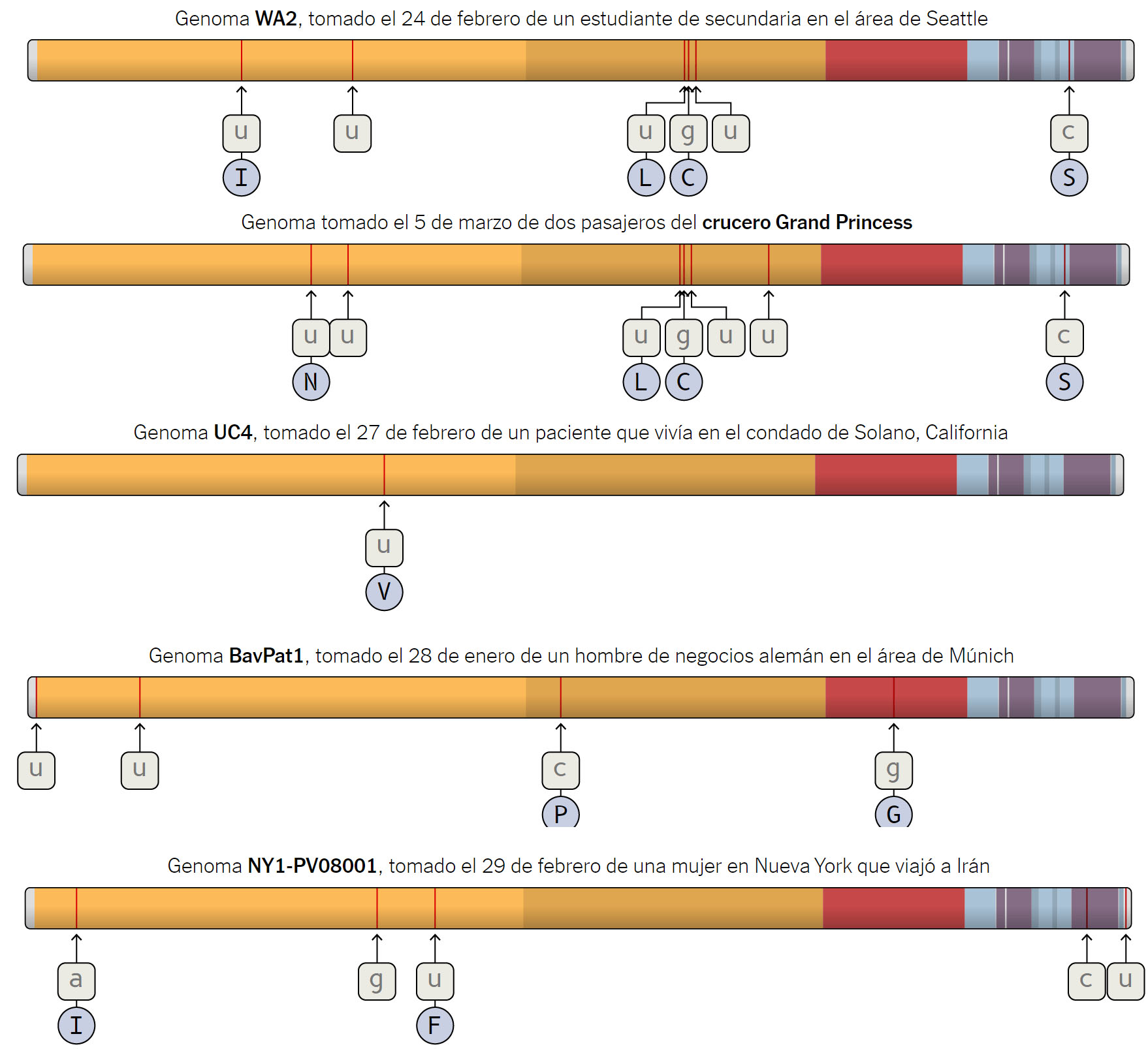
A continuación puedes ver más mutaciones encontradas:
Fuente: The New York Times
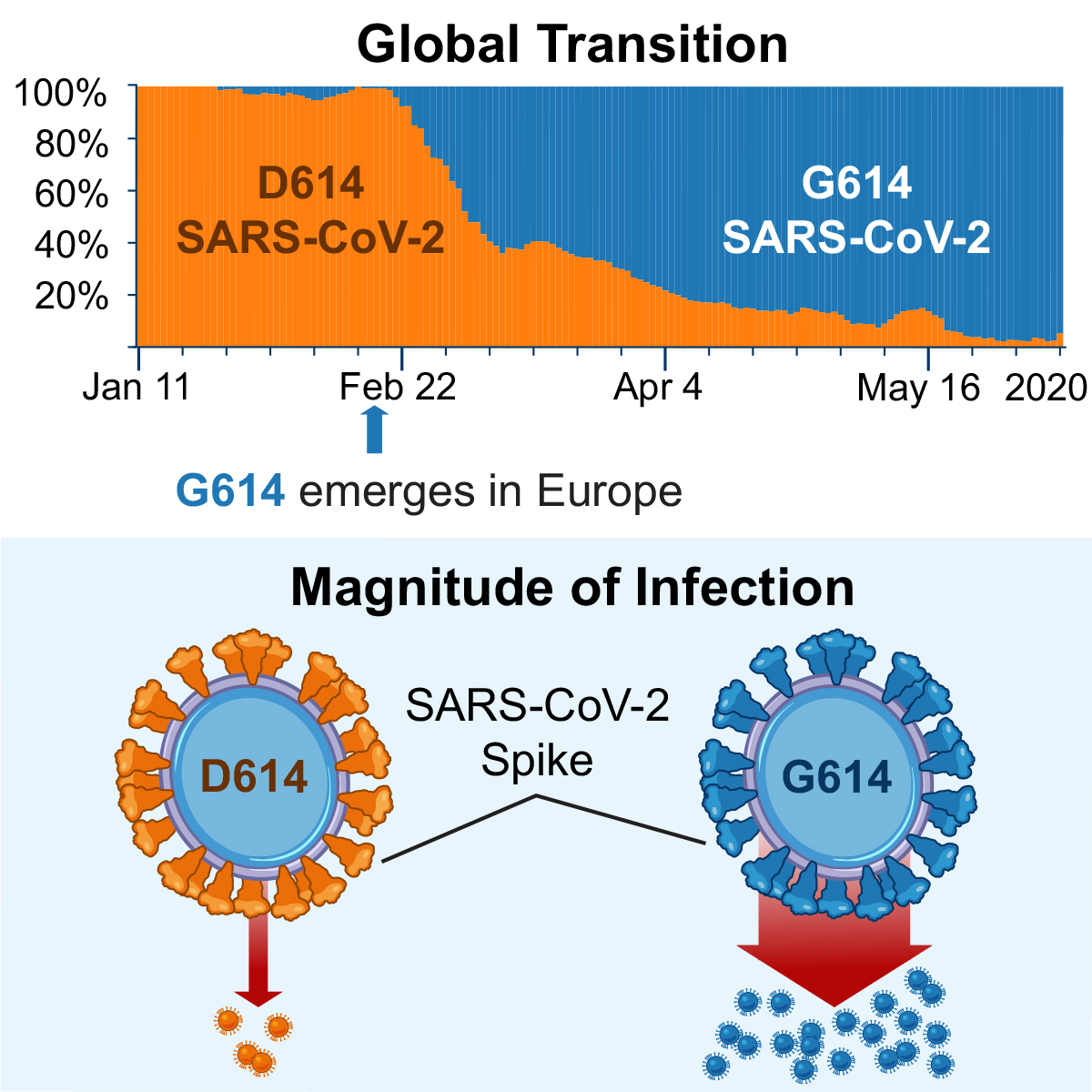
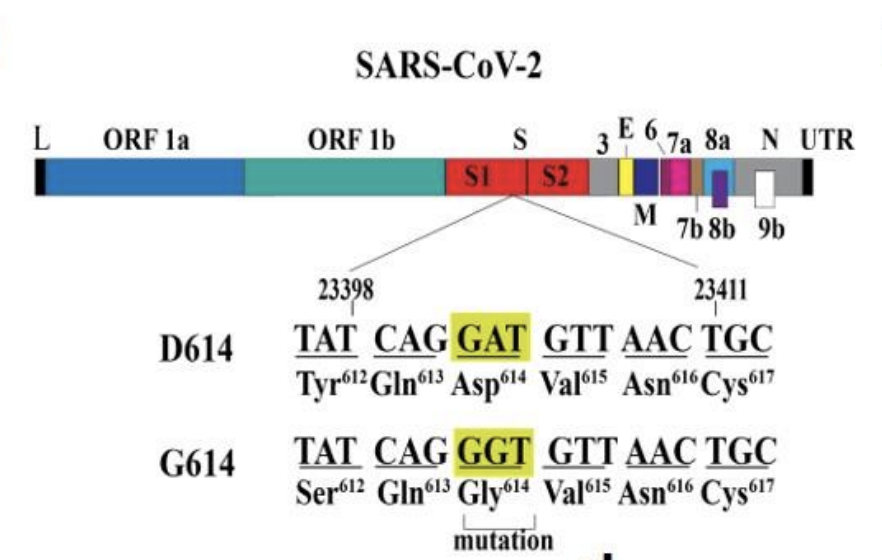
Una mutación significativa fue la mutación D614G. En ella, una de las 30.000 bases nitrogenadas del ARN, según la cepa encontrada en Wuhan, había cambiado en la posición 23.403, donde una Adenina ha cambiado a una Guanina. La ciencia investigaba ahora si esta mínima modificación implicaba una mayor facilidad del virus para contagiar, dado que esta mutación afectaba a la proteína espiga que utiliza el virus para infectar células humanas (ver artículo). Entrando más en detalle, podemos ver que la pandemia se inició con una variante llamada D614 (que tenía el aminoácido ácido aspártico D en la posición 614, y como base nitrogenada adenina), pero se ha acabado imponiendo una mutación, la variante G614 (con aminoácido glicina G en esa posición y Guanina), en lo que se conoce como la mutación D614G, debido al cambio de bases nitrogenadas en el ARN. Para más información, ver Informe del Grupo de Análisis de Coronavirus del ISCII.

Los científicos de todo el mundo han aunado esfuerzos para descubrir cuándo y cómo muta el virus. Ya en enero, investigadores chinos divulgaron la primera secuencia del genoma del virus, y hoy en día, hay decenas de miles de genomas secuenciados por investigadores de todo el mundo, que se comparten en una plataforma web cuya dirección es www.nextstrain.org. Esto ayuda a combatir el virus, al conocer mejor qué es lo que está haciendo.
Durante la pandemia, se han observado distintas informaciones relevantes en este sentido:
- En mayo, científicos del Instituto de Genética del University College of London identificaro unas 200 mutaciones genéticas recurrentes en el coronavirus (ver noticia)
- En mayo, hubo fundadas sospechas en China de mutaciones que hacen al virus más contagioso porque se tarda más en detectarlo (ver noticia)
- En junio, un equipo del Instituto de Investigación Scripps de Florida aseguraba descubrir que una leve mutación genética que incrementaba de manera significativa su capacidad de infección (ver noticia)
- Se sospecha que en Europa y Estados Unidos circula una cepa con una pequeña mutación que lo hace más infeccioso (ver noticia)
También se ha especulado con que alguna mutación lo haya hecho más virulento, y también lo contrario, con que se haya ido atenuando, pero no hay nada confirmado en este sentido. Hasta ahora, la mayoría de las mutaciones encontradas no son significativas, y tampoco se ha descubierto ninguna cepa que sea resistente frente a la inmunidad creada por una infección previa, cosa que sí sucede en otros virus como la gripe. A día de hoy no existe evidencia que D614G o A222V afecten la severidad de la enfermedad o reduzcan la capacidad de las vacunas en ensayos clínicos para proteger contra COVID-19. Sin embargo, un estudio sobre una variante N439K (que cambia asparagina por lisina en la posición 439 de la secuencia de aminoácidos de la proteína espiga) concluye que esta cepa mejora el contacto del virus con la célula que infecta, aunque tiene el mismo resultado clínico que otras, aunque es de interés su vigilancia porque tiene el potencial de evadir la inmunidad previa desarrollada por el individuo (ver estudio).
Hay investigadores que señalan que un virus típico del SARS-CoV-2 puede acumular solo dos mutaciones de un solo nucleótido (una letra) por mes en su genoma, lo que sería una tasa de cambio mucho menor que la de otros virus como la gripe o el VIH. Del mismo modo, en la web www.gisaid.org se han secuenciado y hecho públicos más de 90.000 genomas de virus aislados, de forma que dos virus cualquiera, recolectados desde cualquier parte del mundo, difieren como promedio en solo 10 nucleótidos de un total de 29.903. A pesar de la lenta tasa de mutación que presenta el virus, como la tasa de contagio es elevada, se han catalogado más de 12.000 mutaciones. Muchas de ellas no tendrán consecuencias significativas en la capacidad de contagio o infección del virus, porque no alteran la forma de una proteína, mientras que se cree que aquellas mutaciones que cambian proteínas tienen más probabilidades de dañar su capacidad que de mejorarla.
Además, a finales de julio, un nuevo estudio, publicado en la revista Nature y llevado a cabo por investigadores de la Universidad de Texas, reveló que el virus realiza mutaciones para engañar a las células y que así no lo reconozcan como un agente patógeno (ver noticia). Este hallazgo podría facilitar el camino para el diseño de fármacos específicos contra el SARS-CoV-2, así como otros coronavirus emergentes en un futuro.
A primeros de agosto, un estudio publicado en la revista Frontiers in Microbiology, llevado a cabo por investigadores de la Universidad de Bolonia, basándose en el análisis de 48.635 genomas de coronavirus aislados en laboratorios de todo el mundo, informaba de que el virus presentaba poca variabilidad, mucha menos que el virus de la gripe, y por tanto presentaba un bajo cambio evolutivo, por lo que los tratamientos y vacunas que se estaban elaborando podrían ser efectivos contra todas las cepas del mismo (ver artículo). Decía que había seis cepas conocidas, que clasificó como L, S, V, G, GR y GH:
- La cepa L es la que apareció en diciembre en Wuhan (China)
- La cepa S fue la primera mutación de la original, apareciendo a principios de 2020, y ya detectada en su día en China (ver noticia)
- Las cepas V y G surgieron a mediados de enero de 2020
- La cepa G mutó en las cepas GR y GH a finales de febrero de 2020
Con todo, las cepas G y sus mutaciones GR y GH son las más extendidas, representando el 74% de las cepas analizadas en el estudio, siendo la G y la GR las más frecuentes en toda Europa, si bien en España también se puede encontrar la cepa S. La GH es poco frecuente en Italia pero no así en Francia y Alemania. En América del Norte también estaría más extendida la GH (aunque en EEUU también se encuentra la S), mientras que en América del sur la GR es la predominante. En Asia se han propagado las cepas G, GH y GR desde marzo. Se cree que las cepas L y V desaparecen gradualmente.
En los primeros casos de reinfección que se documentaron en agosto, se pudo constatar que los genomas del virus, en cada una de las infecciones, eran distintos. Por tanto, las distintas mutaciones que pueda haber del virus podrían favorecer los casos de reinfección.
En el tercer trimestre de 2020 se identificó una cepa desconocida en Noruega. El 22 de septiembre de 2020, se confirmó un positivo entre los pasajeros en un viaje en autocar en el sur de Noruega. Casi todos los pasajeros fueron infectados durante los siete días que viajaron juntos. Se rastrearon contactos estrechos, llegándose a confirmar un centenar de positivos al menos, incluyendo cinco hospitalizaciones. El Instituto Noruego de Salud Pública examinó el material genético del coronavirus de varios casos y detectó una variante del virus con un cambio en la proteína espiga. Era la primera vez que se observaba esa mutación en aquel país, y no se sabía cómo pudo llegar, aunque sí se han visto cepas casi idénticas en otros países europeos, perteneciente a un subgrupo genético del coronavirus llamado B.1.160. No se tenía constancia de que la enfermedad con esta mutación fuese más grave, aunque podría afectar a su infectividad, pues se vio que muchos contactos cercanos fueron infectados, por lo que la mutación podría contribuir a una mayor transmisión (ver noticia).
A finales de octubre, un estudio sugería que la variante S614G se habría hecho dominante, con más afinidad por los receptores ACE2, y con capacidad para replicarse más en células de vías respiratorias (ver estudio).
En noviembre, el gobierno danés anunció el sacrificio de miles de visones, pues se detectó en estos animales una mutación del virus que podía comprometer las vacunas ya en avanzada fase de desarrollo. Al parecer, esta variante que también se había detectado en humanos (al menos en 214 personas a primeros de noviembre, aunque aumentando en número y en extensión geográfica), no se veía afectada por los anticuerpos producidos por pacientes recuperados de COVID (ver noticia), aunque otras informaciones apuntan a que si bien es neutralizado de forma menos eficaz cuando el número de anticuerpos es bajo o medio, sí que es neutralizado con altos niveles de los mismos, y además no tendría ventajas a la hora de replicarse, por lo que la orden de eliminación de estos animales estaba destinada a prevenir el desarrollo de cepas cada vez más divergentes (ver informe). Según un informe del Statens Serum Institut (SSI) –organismo danés que controla las enfermedades infecciosas–, pruebas de laboratorio han servido para detectar que la cepa presenta mutaciones significativas en la proteína espiga, que es la proteína a la que deben adherirse los anticuerpos para inactivar el virus. Esta nueva variante, al alterar esta proteína clave, podría poner en riesgo la efectividad de las vacunas en desarrollo, por lo que se tomó la decisión de sacrificar a estos animales. En España, en julio, se sacrificaron también cerca de 100.000 visones en Aragón (ver noticia), habiendo sido positivo al menos 7 trabajadores de la explotación, aunque se desconoce si hay relación entre este caso y el caso danés. Sobre la mutación en virus de visones, al parecer existen varias variantes, definidas por una mutación denominada A22920T (al cambiar adenina por timina en la posición 22920 del ARN) y también Y453F (por el cambio de aminoácido tirosina a fenilalanina en la espícula), y adicionalmente otras mutaciones en algunos casos como, S1147L y M1229I, o una deleción denominada 69-70delHV (mutación genética en la cual se pierde material genético). Además, según algunas informaciones, podrían también contar con la mutación N439K mencionada anteriormente.
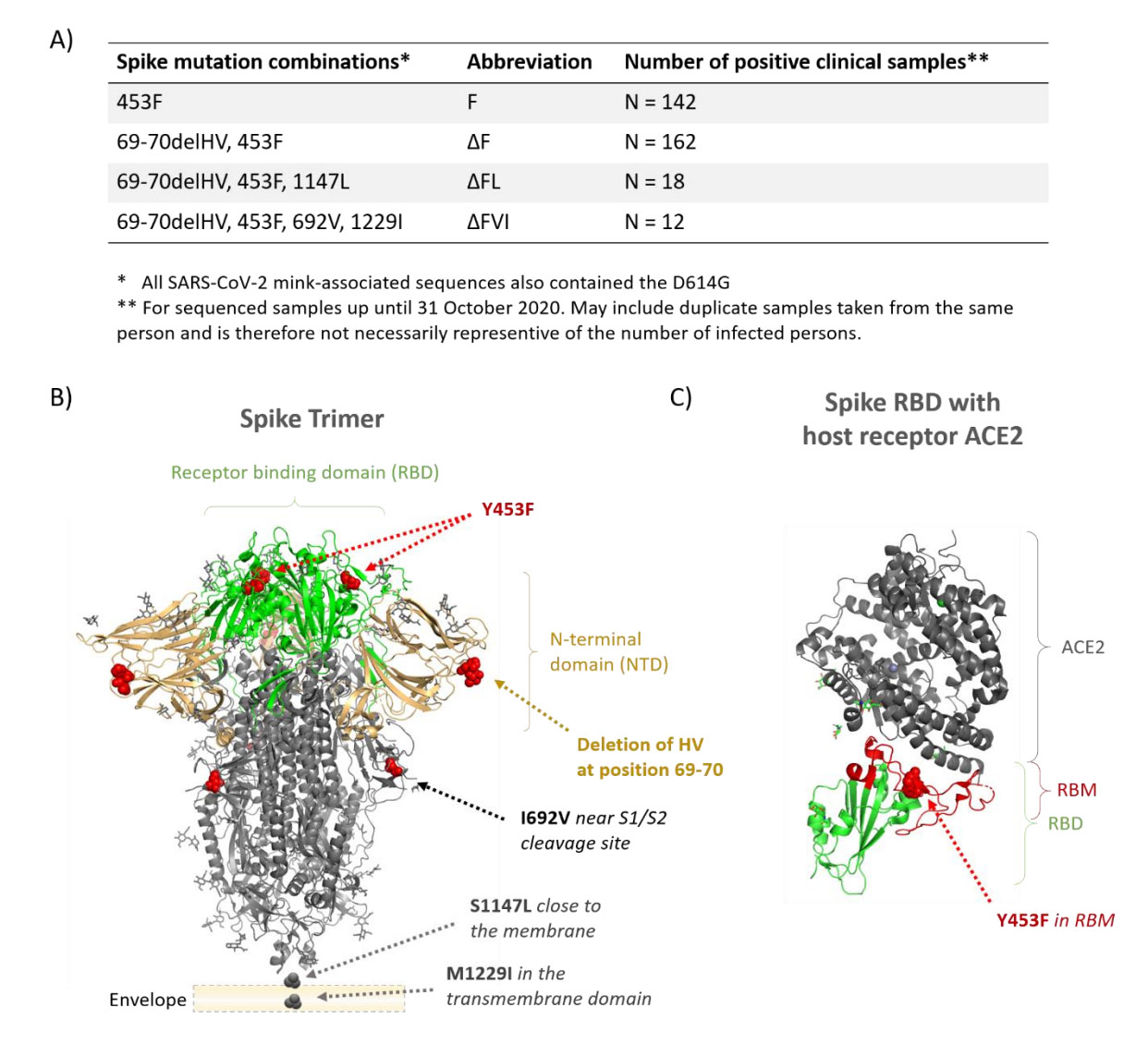
Fuente: Informe sobre los casos de visones (ver informe)

Fuente: sciences et avenir
En diciembre se supo que una especie de visón silvestre, en Utah, había sido contagiado por el SARS-CoV-2, tras confirmarse mediante PCR. Se especulaba con que podía haber sido infectado por algún ejemplar que había huido de una granja de visones. Esta situación preocupa especialmente, pues el virus podría mutar de distinta forma si se propaga en otros animales, y volver más adelante a los humanos a través de otro caso de zoonosis, de forma que una eventual nueva cepa mutada pudiera esquivar la inmunidad desarrollada en humanos previamente vía infección o vacuna (ver artículo).
El hecho de que el virus pueda expandirse en otros animales y mutar en ellos es una preocupación con fundamento, más cuando se han identificado alrededor de sesenta especies susceptibles a la infección por SARS-CoV-2, como son murciélagos, gatos, leones, hurones, perros, tigres, visones, mapaches, gorilas o hamsters, si bien algunos otros parecen no susceptibles, como por ejemplo ratas, cerdos y jabalíes (ver artículo).
Desde diciembre de 2020, proliferaron las variantes de interés (VOI) y las variantes de preocupación (VOC). Algunos estudios sugieren que ciertas mutaciones de las VOC podrían escapar de los anticuerpos generados por vacunas o infección previa, si bien también es posible que el impacto sobre las células T generadas sea insignificante, como sugiere un preprint publicado en marzo de 2021 (ver estudio).
Puedes seguir aquí todo lo relativo a las VOC y VOI.
VARIANTE/CEPA de KENT, B.1.1.7 O 501Y.V1, ALFA (REINO UNIDO)
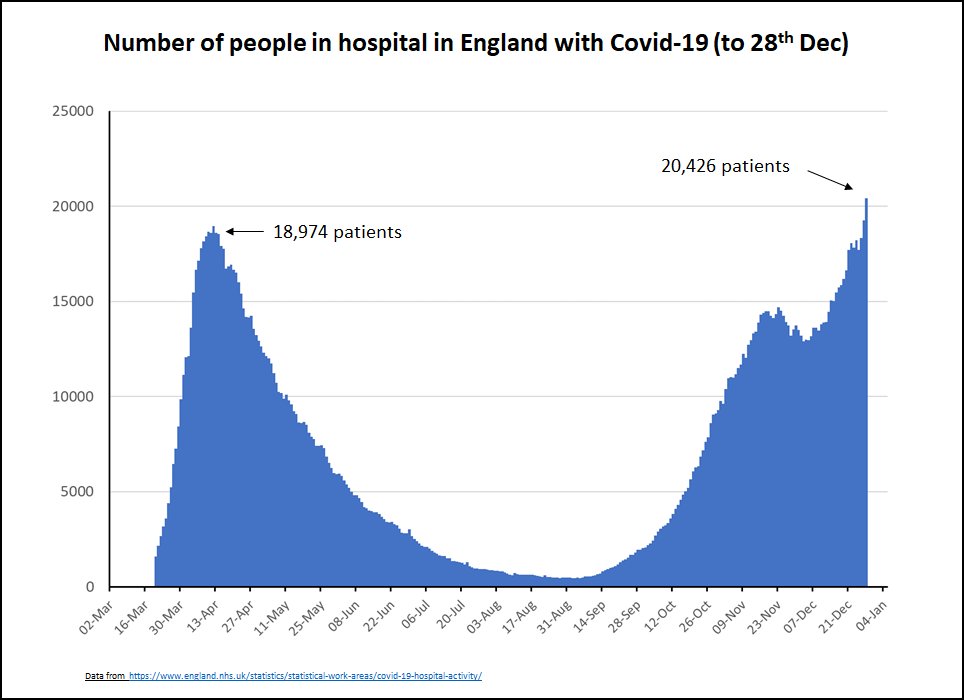
En la segunda quincena de diciembre de 2020, el Reino Unido alertaba de una nueva mutación que parecía estar propagándose más, lo cual también preocupaba de cara a la efectividad de las vacunas (ver noticia). El gráfico de hospitalizaciones en el Reino Unido, que alcanzó un máximo en diciembre, superior a la ola de primavera de 2020, podría ser un indicador de su mayor contagiosidad. Un estudio publicado en febrero 2021, realizado en Reino Unido, destacaba que no se había encontrado evidencia de que haya ocurrido una enfermedad más grave en niños y jóvenes durante la segunda ola (invierno) allí, lo que sugiere que la infección con la variante B.1.1.7 no da como resultado un curso clínico apreciablemente diferente al de la cepa original, por lo que se supone que el COVID-19 respiratorio agudo severo sigue siendo una ocurrencia poco común en niños y jóvenes (ver estudio).
Y a finales de diciembre, viendo la progresión en distintas zonas de Reino Unido, se pensaba que las infecciones por la nueva variante podrían representar más del 90% de los nuevos contagios en febrero.
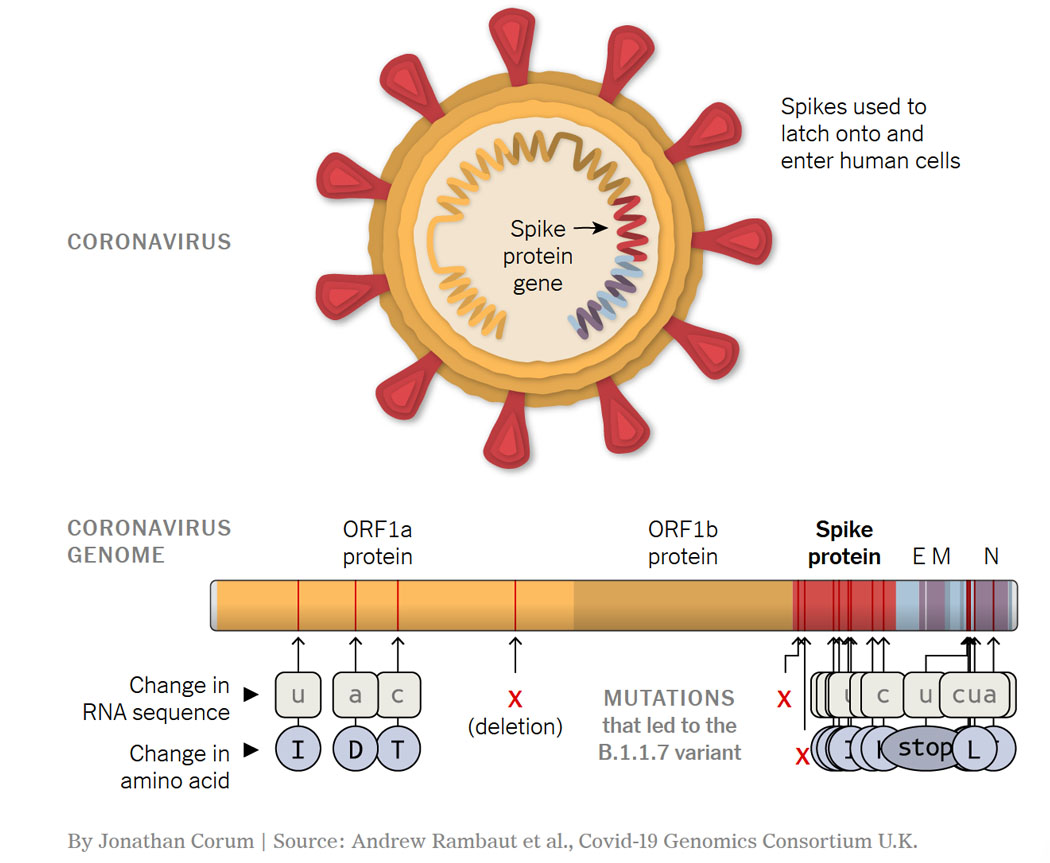
Fuente: The New York Times
Fuente: The New York Times
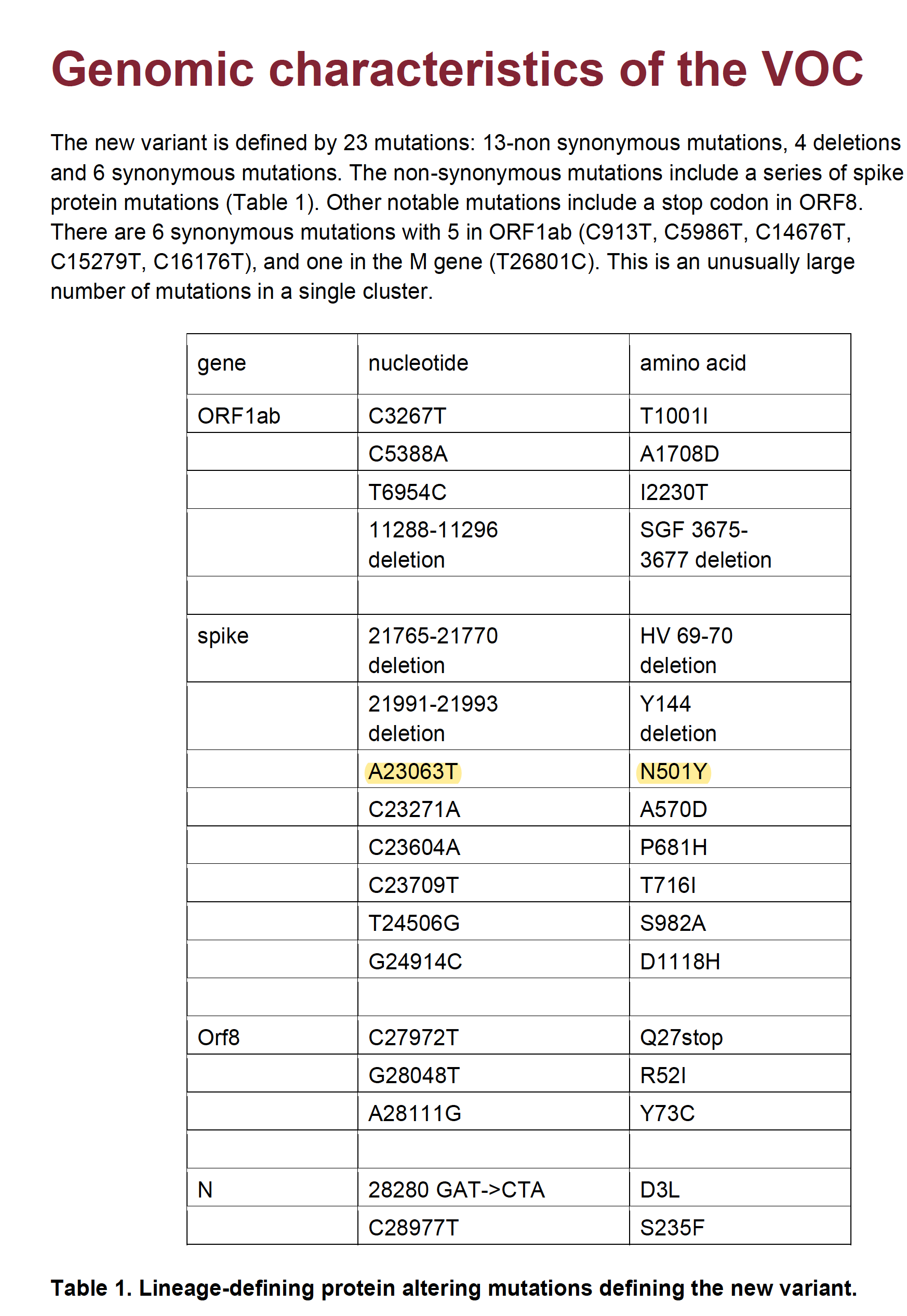
Mutaciones de la variante
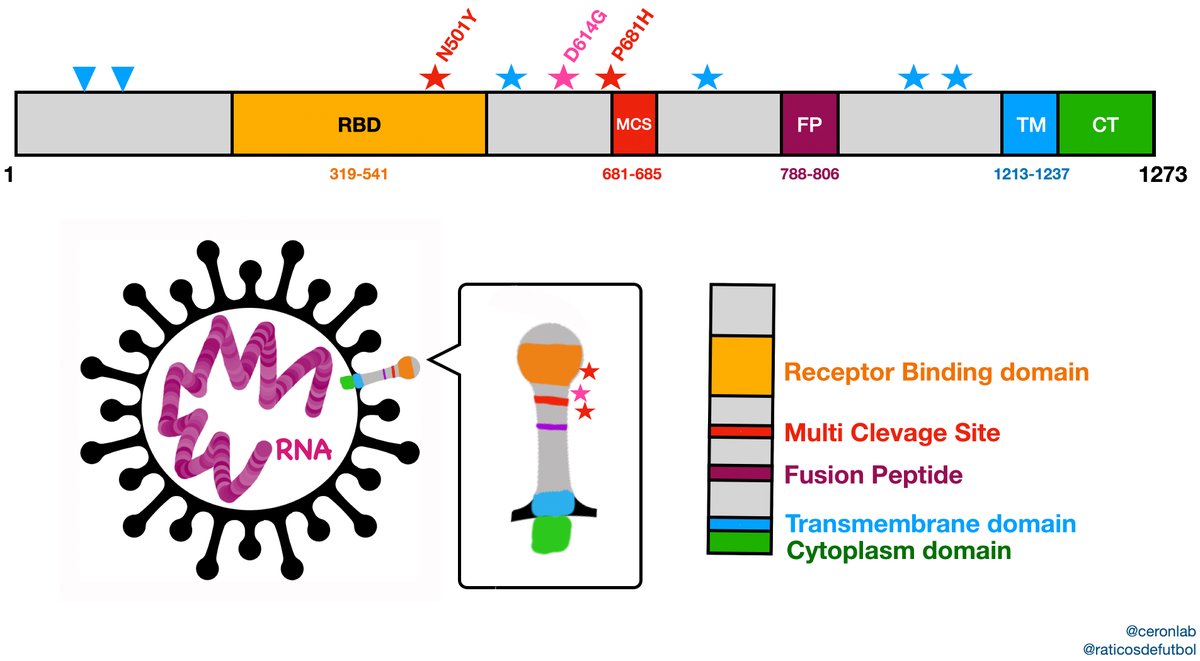
Más concretamente, lo que parecía preocupar más a algunos científicos era la combinación de 3 de estas mutaciones, porque podrían mejorar la transmisión: la D614G, la P681H, que se ubica en una zona de corte de proteasa, requerida para la entrada a las células, y la comentada N501Y, que dentro de la proteína espiga, afectaba a la región denominada RDB (Receptor Binding Domain), que es la que se une a los receptores ACE2, y que es a la que se cree que se unen los anticuerpos para su neutralización. Cada mutación aparece 3 veces en la espiga, pues cada espiga es la unión de tres proteínas S.
Resumiendo, las mutaciones que contiene tendrían los siguientes efectos:
- N501Y ayudaría a que el virus se adhiera con más fuerza a las células humanas, aunque no parece probable que ayude al virus a evadir las vacunas actuales.
- P681H , que puede ayudar a las células infectadas a crear nuevas proteínas de pico (S) de manera más eficiente.
- Las deleciones H69-V70 e Y144 / 145 alteran la forma del pico y pueden ayudarlo a evadir algunos anticuerpos.
Con respecto a su procedencia, se especula con que se haya originado en personas en las que el virus no se haya eliminado por completo en poco tiempo. En una infección típica, las personas contraen el coronavirus y se vuelven infecciosas durante unos días antes de mostrar síntomas. Poco a poco el virus luego se vuelve menos abundante en el cuerpo a medida que el sistema inmunológico se va defendiendo, hasta ser eliminado por completo en pocas semanas, excepto en aquellos casos graves. Si el virus ataca a personas inmunodeprimidas, el virus puede permanecer meses, y este lapso de tiempo ampliado puede ser favorable a que vaya acumulando más mutaciones. Otras teorías serían que la variante podría haber sido impulsada adicionalmente por medicamentos administrados a estos pacientes (al hacerse resistentes a fármacos como los anticuerpos monoclonales), o que podría haber obtenido nuevas mutaciones al propagarse a través de una población animal, como visones, antes de volver a entrar en la población humana.
La mayoría de los expertos dudan de que tenga un gran impacto en las vacunas, aunque todavía no es posible descartar ningún efecto. Es concebible que una mutación pueda cambiar la forma de la proteína S, haciendo más difícil que los anticuerpos los controlen, y las mutaciones de B.1.1.7 incluyen ocho en esta proteína, pero nuestro sistema inmunológico puede producir una variedad de anticuerpos contra una sola proteína viral, por lo que es menos probable que los virus puedan escapar fácilmente de su ataque. Por tanto, el impacto de estas mutaciones con respecto a las vacunas debería analizarse caso a caso. Los anticuerpos podrían ser efectivos contra distintas variantes de la proteína S si las mutaciones no son lo suficientemente significativas o grandes, y de hecho esto es lo que se espera. En el caso de la mutación de Reino Unido, la farmacéutica Moderna confiaba en que su vacuna siguiera siendo efectiva. No obstante, existe un estudio que trataba de evaluar si los coronavirus evolucionan para escapar de la inmunidad y, de ser así, con qué rapidez, que se centró en la evolución histórica del coronavirus humano 229E. En dicho estudio se identificaron sueros humanos de las décadas de 1980 y 1990 que tenían anticuerpos neutralizantes frente a 229E (comparables a los anticuerpos de SARS-CoV-2 inducidos por la infección o vacunación). Se probaron estos sueros contra cepas 229E aisladas que aparecieron años más tarde de la recolección de sueros (cepas más nuevas, por así decirlo), y se encontró el hallazgo de que esos anticuerpos neutralizantes eran menos efectivos contra esos virus que habían aparecido más tarde, llegando a darse en algunos casos la no neutralización de cepas aisladas entre 8 y 17 años después de la toma de la muestra. Parece que la disminución de la neutralización de cepas más nuevas se debía a la evolución antigénica de la proteína espiga, por lo que si se extrapola este resultado al SARS-CoV-2, podría ser necesaria una actualización periódica de las vacunas (ver estudio). También en este sentido, Novavax informaba a finales de enero de 2021 que su vacuna era algo menos eficaz contra esta variante, aunque la eficacia seguía siendo alta (83% frente a 90%) (ver noticia).
El 22 de enero de 2021 se publicaba un preocupante informe sobre esta variante realizado por expertos del Reino Unido (ver informe). La variante era más transmisible y se había hecho dominante en la mayor parte del Reino Unido, y podría estar incluso asociada con una mayor mortalidad, que se cifraba en torno a un 30 a 40% más.
En los primeros días de febrero de 2021, se detectó un hallazgo altamente preocupante. Al parecer, se habían detectado 11 casos de la variante del Reino Unido (B.1.1.7) que también incluían la mutación E484K (la que se encuentra en las denominadas variantes de Sudáfrica y Manaus). Esta mutación se había asociado con un alto nivel de escape del virus a anticuerpos neutralizantes de otras variantes, lo que podría suponer también una pérdida de eficacia de las vacunas disponibles en ese momento (ver dossier). En marzo de 2021, se detectó una variante similar en Oregon, Estados Unidos (ver noticia).
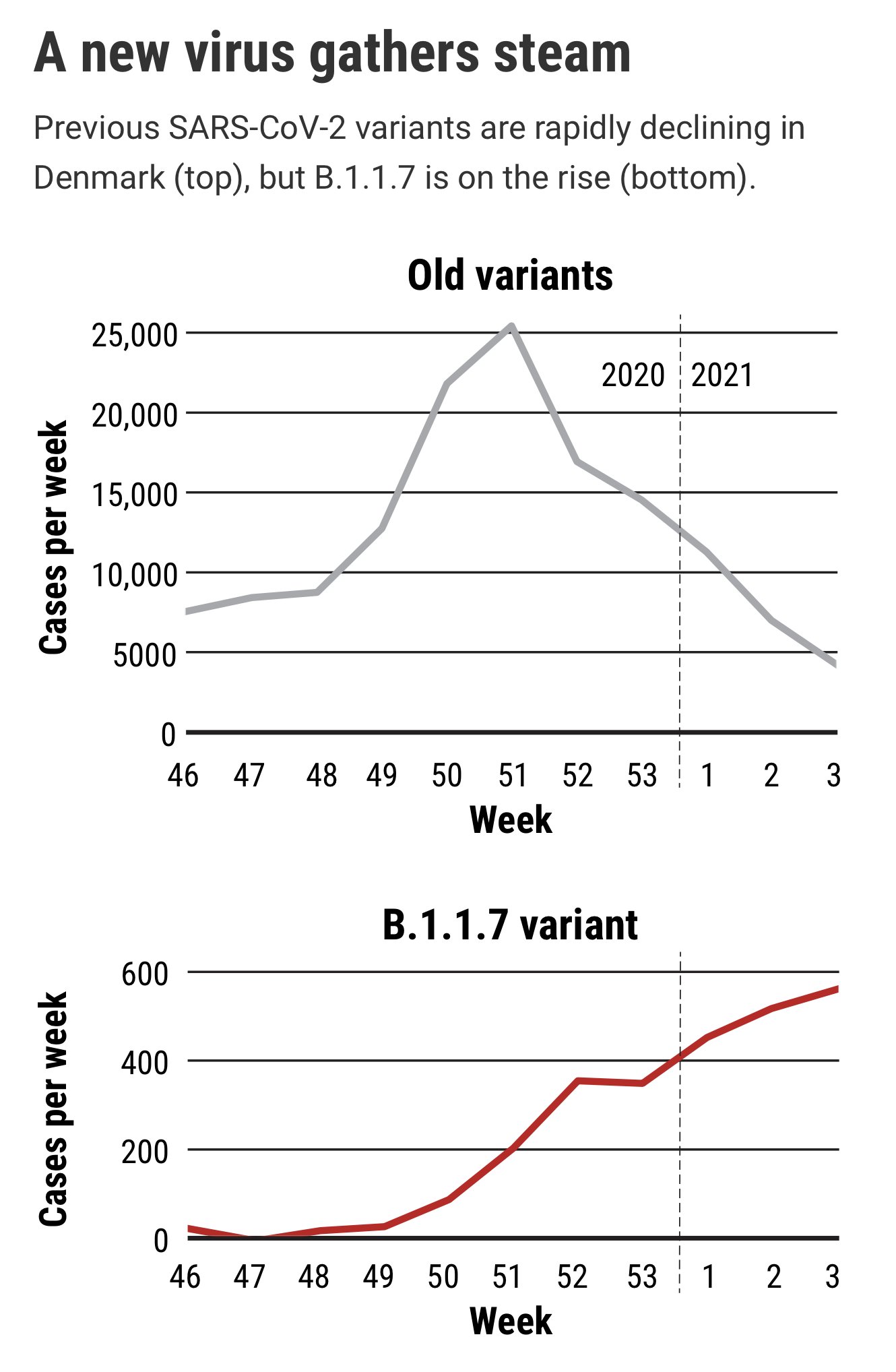
Su contagiosidad llevó a que en Ontario, Canadá, se asegurara que podría bastar tan solo 1 segundo sin mascarilla para provocar el contagio (ver noticia). También se pudo comprobar en este mes cómo fue ganando terreno en Dinamarca; en este país, donde se secuencian muchas variantes, se observaba que tras unas semanas de confinamiento en todo el país, las cifras de contagios diarios cayeron de más de 3000 a mediados de diciembre de 2020, a unos pocos de cientos a primeros de febrero. Sin embargo, lo preocupante es que se observaban como dos epidemias: una, que se reduce rápidamente, causada por variantes más antiguas de SARS-CoV-2, y un brote más pequeño y de crecimiento lento de la variante B.1.1.7. Se creía que si esta última sseguía extendiéndose al mismo ritmo en Dinamarca, se convertirá en la variante dominante a finales de febrero y haría que el número total de casos aumentara de nuevo (ver artículo).
Fuente: Science
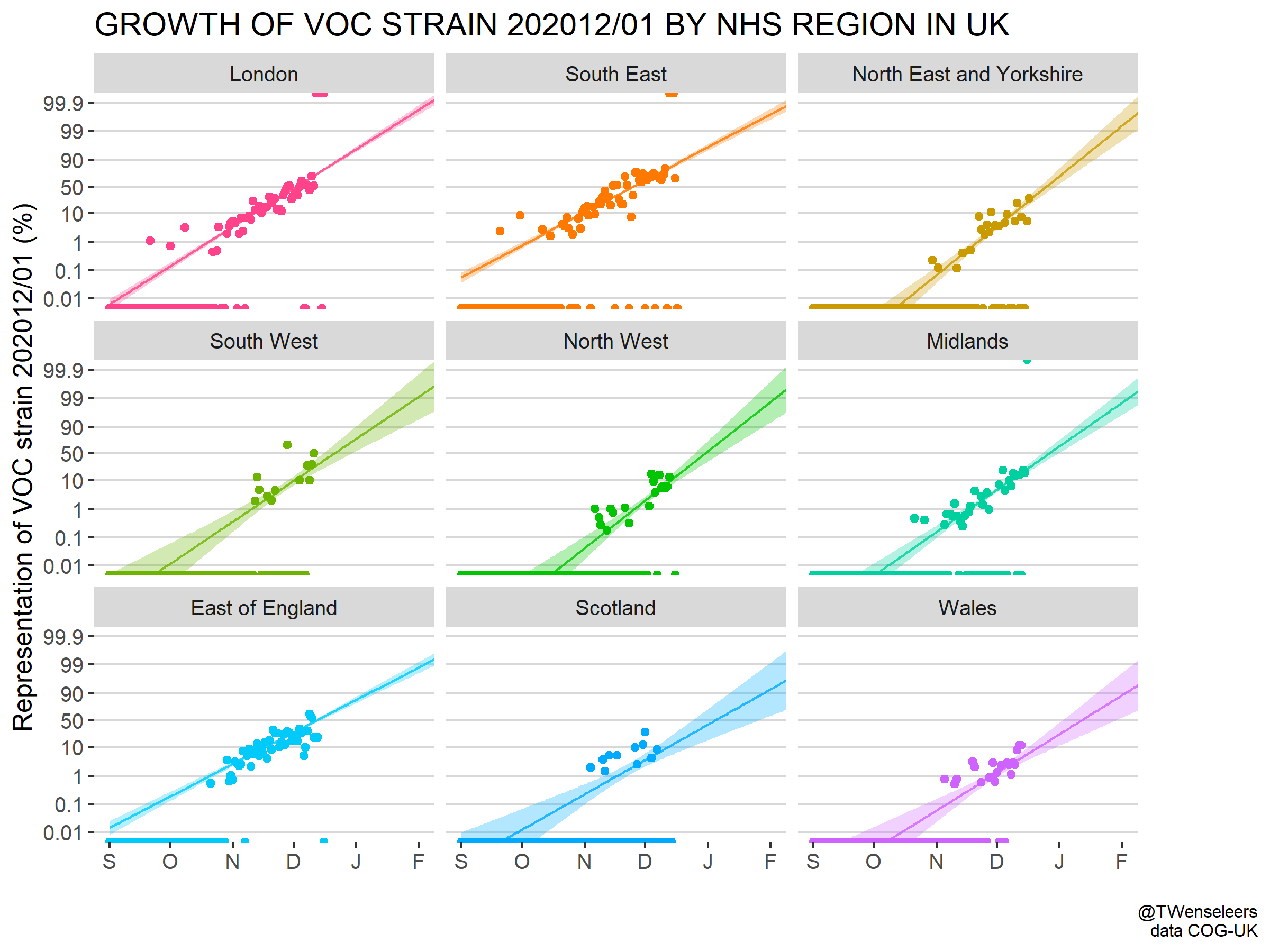
A primeros de marzo de 2021, ya se veía en España como la tendencia es que esta variante se estaba convirtiendo en dominante, subiendo claramente en el porcentaje de contagios sobre el total.
Fuente: Miguel Ángel Reinoso
En mayo de 2021 se pubicó un estudio que analizó diversos brotes de la variante inglesa (B.1.1.7) en centros infantiles de Alemania, concluía que era factible que los niños y adultos tengan una probabilidad similar de infectarse, así como de transmitir el virus a las personas en su domicilio (ver estudio).
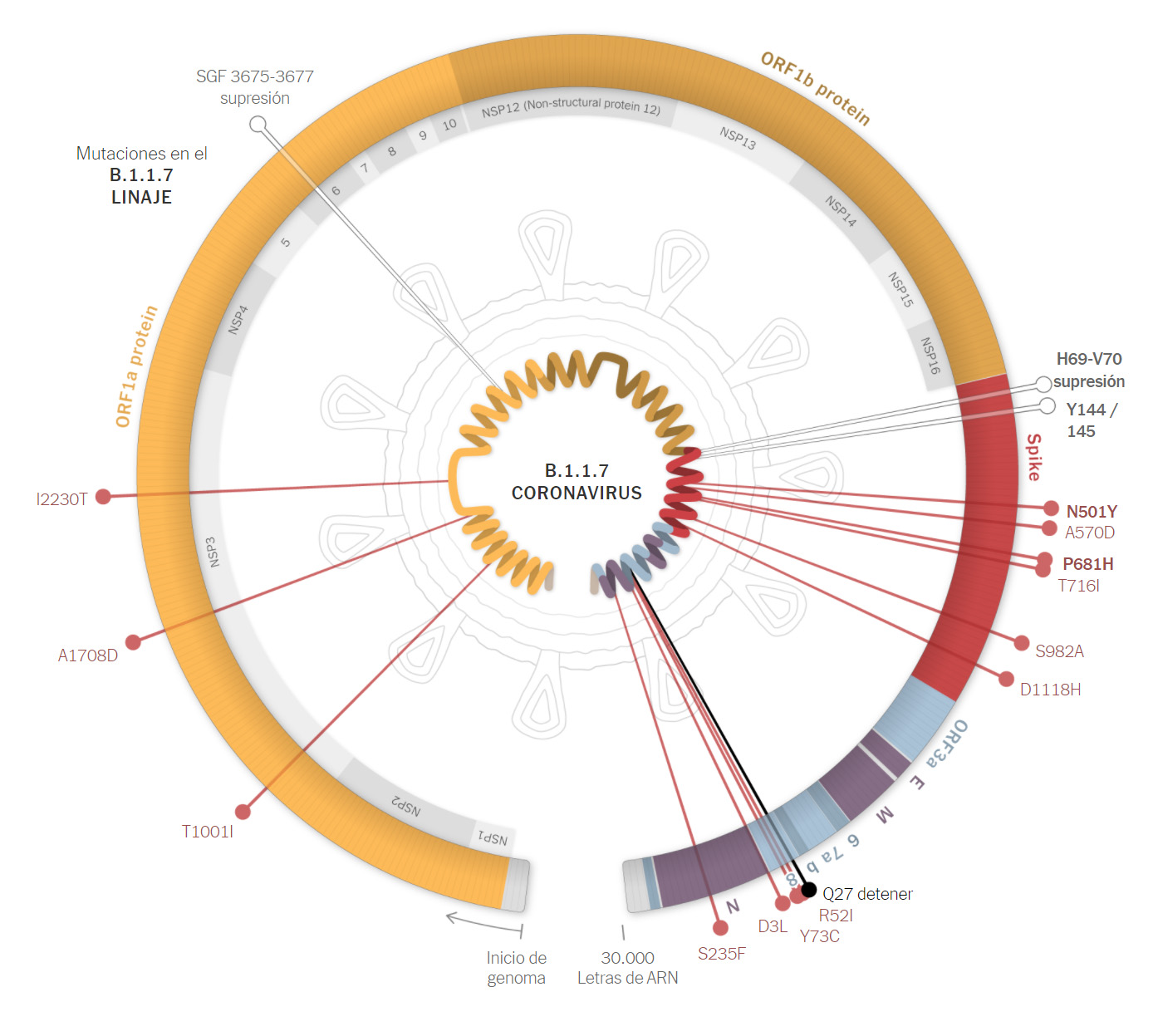
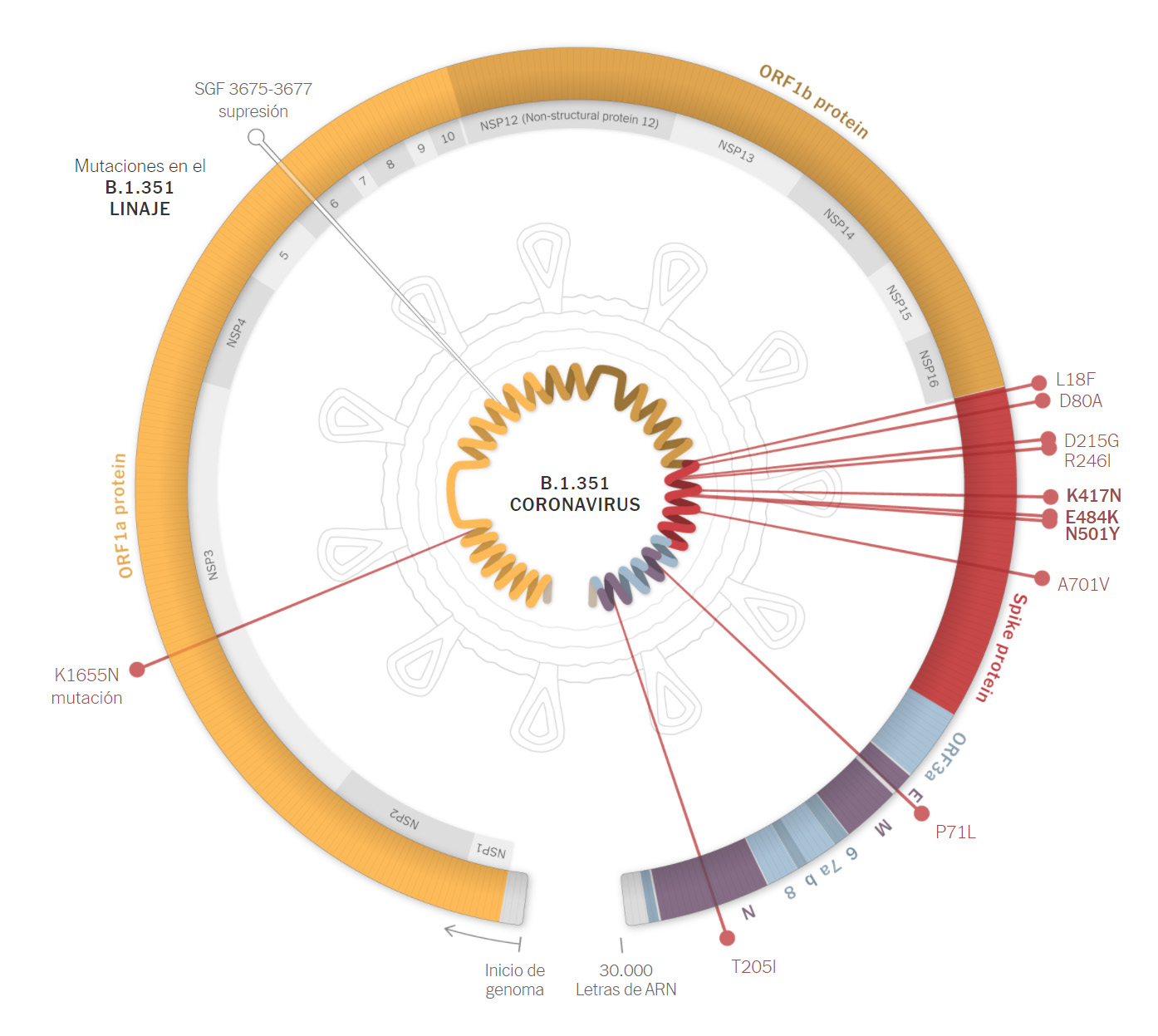
VARIANTE/CEPA 501Y.V2 o B.1.351 (SUDÁFRICA)
En realidad, en diciembre de 2020, había dos nuevas variantes identificadas, que incluían la mutación N501Y en la espícula del virus; la mutación B.1.1.7 en Reino Unido, y la 501Y.V2 en Sudáfrica, que cambian asparagina por tirosina en la posición 501 de la proteína S. Ambas, con aparente fuerza para su transmisión, no son exactamente la misma variante, pues han surgido de forma separada, y tienen otras mutaciones en otras posiciones que las distinguen (también en la proteína S), y además, se ha documentado que esta mutación ha ocurrido varias veces en todo el mundo, si bien no todas se han mostrado tan prominentes. La variante de Reino Unido además se había detectado pronto en Gales, Escocia, Irlanda del Norte, Australia y Dinamarca, y días más tarde en Italia o Gibraltar, hasta que el 26 de diciembre se confirmó su presencia en Francia y España, probablemente exportadas del Reino Unido, pero se creía que podría estar en algún lugar más de Europa, pues Reino Unido y Dinamarca son los países más prolíficos a la hora de secuenciar genomas dentro del continente. Incluso por esto mismo, se cree que su origen podría estar en otro país, descubriéndose antes en el Reino Unido por ser uno de los que más muestras de virus secuencian. Análogamente podría pasar con la variante solo detectada en Sudáfrica en ese momento, y que días más tarde se detectó también en el Reino Unido. Las muestras de Reino Unido se habían recopilado en pacientes ya en el mes de septiembre, y cuando los investigadores la vieron de cerca, se sorprendieron por la cantidad relativamente grande de mutaciones en comparación con los casos anteriores, 23 en total, que había adquirido. Estas mutaciones parecían afectar potencialmente a la forma en la que se propaga el virus, pues se estaba propagando rápidamente en partes del sur de Inglaterra, desplazando a otras variantes más dominantes durante meses. Neil Ferguson, epidemiólogo del Imperial College de Londres, estimaba que la variante tenía una tasa de transmisión entre un 50 y un 70% mayor que otras variantes que circulaban por el Reino Unido. A finales de diciembre, se especuló con que esta variante fuera consecuencia de otra variante descubierta en Brescia (Italia) a principios de agosto (ver noticia).
Un estudio confirmó que las vacunas de Pfizer y Moderna generaban anticuerpos similares entre sí, y similares a los que se producen en las personas infectadas (ver estudio), si bien ponía de relieve una menor capacidad neutralizante tanto de los anticuerpos generados por la infección, como por los generados a partir de la vacunación, ante las nuevas variantes detectadas desde diciembre de 2020. En concreto, las mutaciones peligrosas en este sentido son:
- E484K
- N501Y
- La combinación K417N:E484K:N501Y
Fuente: The New York Times
Las mutaciones que contiene tendrían los siguientes efectos:
- N501Y ayudaría a que el virus se adhiera con más fuerza a las células humanas, aunque no parece probable que ayude al virus a evadir las vacunas actuales.
- K417N, que ayudaría a que el virus se una con más fuerza a las células humanas.
- E484K, que puede ayudar al virus a evadir algunos tipos de anticuerpos.
También en este sentido, Novavax informaba a finales de enero de 2021 que su vacuna era algo menos eficaz contra esta variante, entre un 50 y 60%, lo cual no era una buena noticia, porque perdía efectividad frente a otras variantes (superiores al 80%), pero tampoco muy mala noticia, pues presentaba cierto grado de eficacia contra esta preocupante variante (ver noticia). Un estudio de febrero 2021 sugería también que la vacuna de AstraZeneca no parece resultar eficaz frente a la variante sudafricana a la hora de prevenir la COVID19 leve-moderada, aunque quedaría conocer su efecto frente a la enfermedad grave (ver estudio).
Esta variante empezó a ser detectada a primeros de 2021, en España, concretamente en enero en Galicia (ver noticia) y en Cataluña en febrero (ver noticia). Un estudio preliminar publicado en febrero sugería que esta variante, al igual que la brasileña, fue parcialmente resistente a anticuerpos utilizados para tratar el COVID (Casirivimab), o completamente (Bamlanivimab), y fue menos eficazmente inhibida por el suero / plasma de individuos convalecientes o vacunados con la vacuna de Pfizer (ver estudio).
Un estudio realizado en ratones confirmaba que las variantes B.1.1.7 (británica) y B.1.351 (sudafricana) son altamente virulentas en ratones transgénicos K18-ACE2 y muestran patrones patogénicos distintos a los de las primeras cepas del SARS-CoV-2, siendo hasta 100 veces más letales que el SARS-CoV-2 original con la mutación 614D. Estos ratones presentaban lesiones más graves en órganos internos que los infectados con versiones anteriores del virus, presentando depósitos significativos de dímero D en órganos vitales. Los ratones con inmunidad previa por una infección o una inmunización anteriores fueron resistentes a la reinfección de B.1.1.7 o B.1.351, a pesar de tener títulos de neutralización reducidos contra estas VOC (ver estudio).
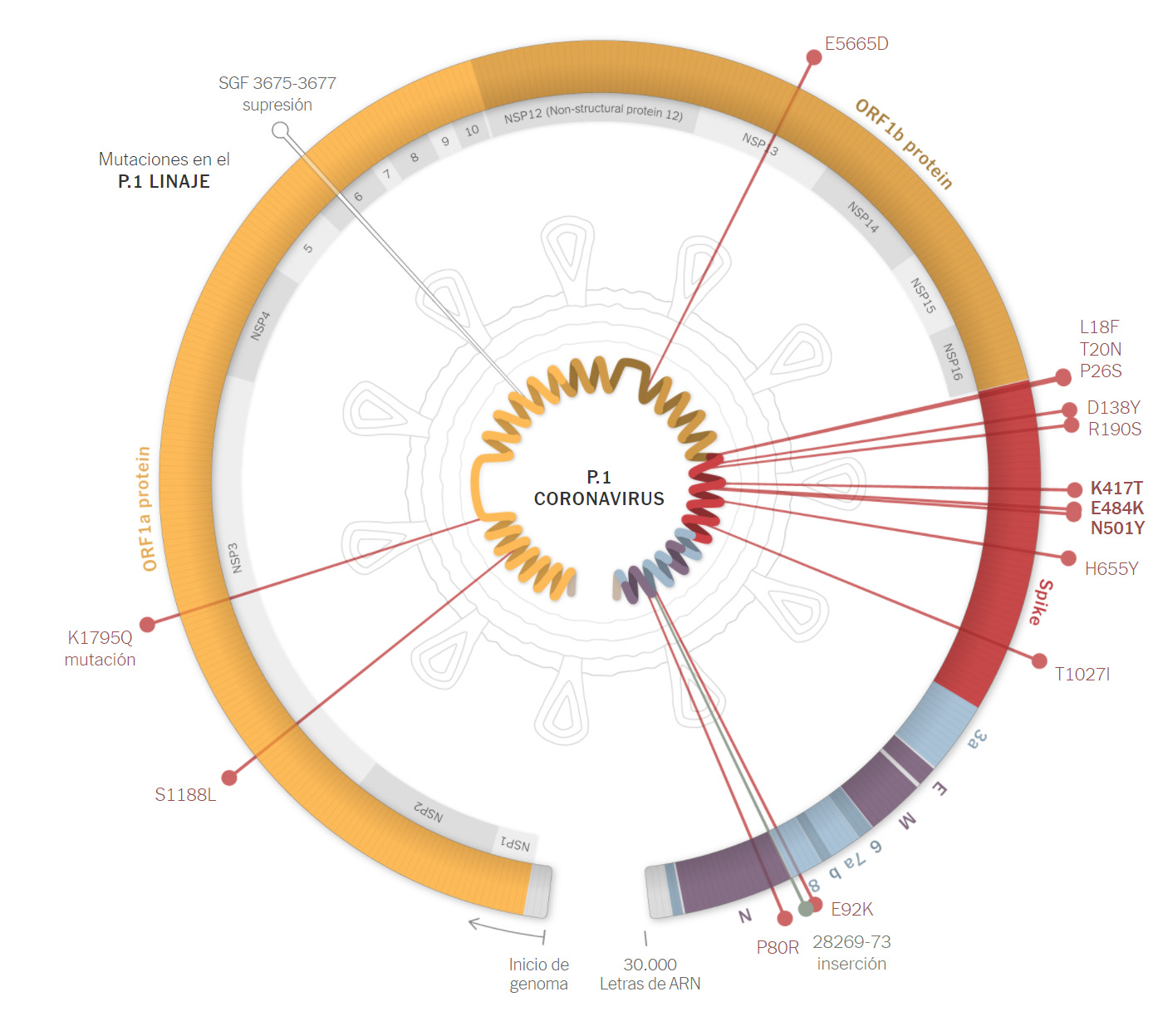
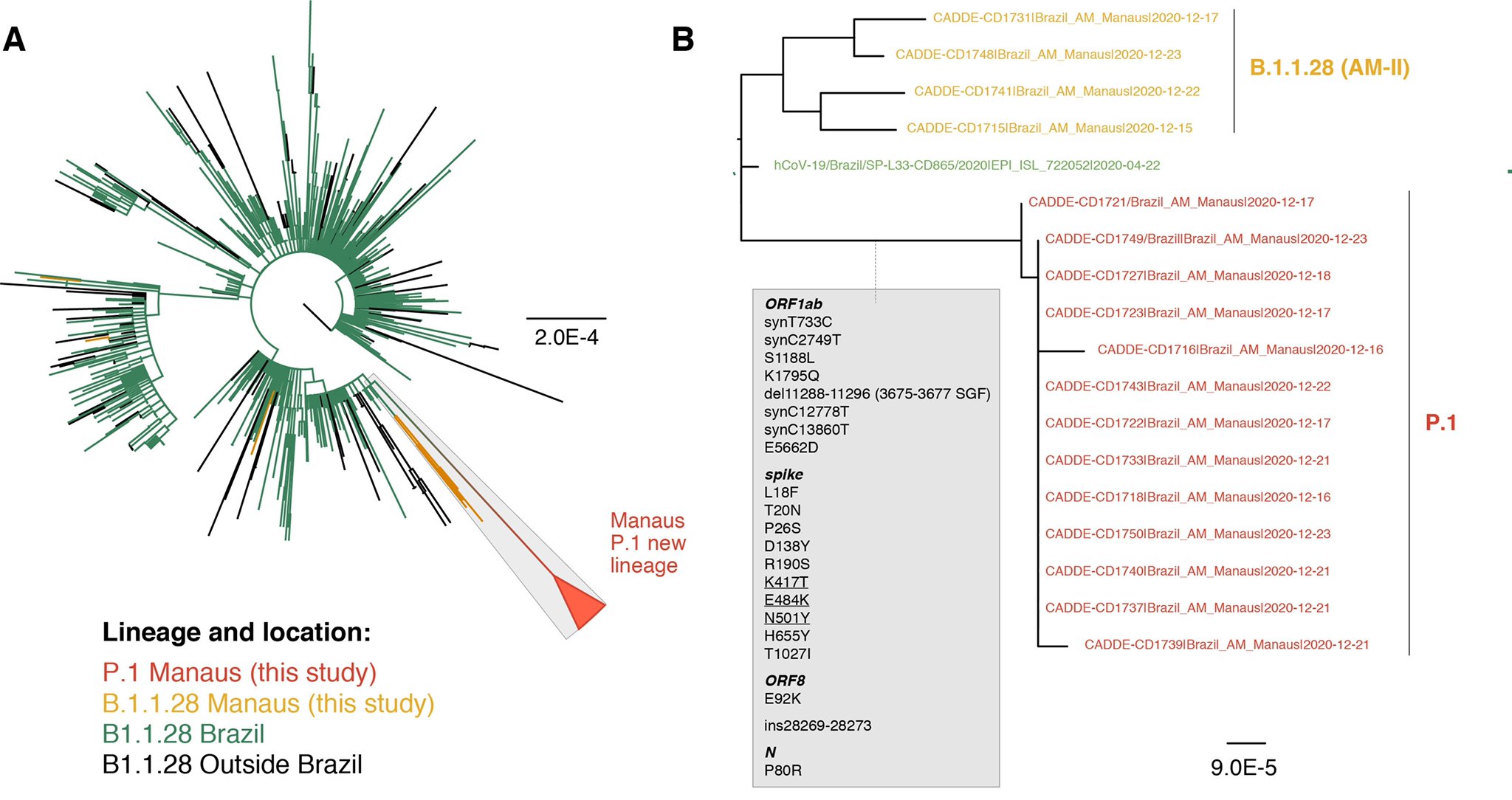
LINAJE B.1.1.28 o P.1 (Manaos, BRASIL)
En octubre de 2020, se calculó que en la ciudad brasileña de Manaos se había infectado sobre el 75% de la población, lo que sugería que se podía estar cerca de la inmunidad de rebaño (ver noticia) (ver artículo). Sin embargo, de repente, la mortalidad empezó a aumentar significativamente de nuevo en enero de 2021, tras un período de relativa calma. Además, la población de Manaos es más joven que en otros lugares (ver noticia). El problema parece ser la variante B.1.1.28/P1, con varias mutaciones en la proteína espiga S, incluyendo la preocupante mutación E484K, que puede evadir anticuerpos, además de la K417N y la N501Y. Además, hay otra variante, denominada B.1.1.28, que también tiene la mutación E484K. Además se han documentado casos de reinfección con variantes que incluyen esta mutación. También fue encontrada en Japón.
Fuente: The New York Times
Las mutaciones que contiene tendrían los siguientes efectos:
- N501Y ayudaría a que el virus se adhiera con más fuerza a las células humanas, aunque no parece probable que ayude al virus a evadir las vacunas actuales.
- K417T, que ayudaría a que el virus se una con más fuerza a las células humanas, análogamente a la K417N de la variante sudafricana.
- E484K, que puede ayudar al virus a evadir algunos tipos de anticuerpos.
La mutación en E484K podría suponer una reducción de hasta 10 veces la capacidad de neutralización de anticuerpos previos, según algunas fuentes.
Un estudio preliminar publicado en febrero sugería que esta variante, al igual que la sudafricana, fue parcialmente resistente a anticuerpos utilizados para tratar el COVID (Casirivimab), o completamente (Bamlanivimab), y fue menos eficazmente inhibida por el suero / plasma de individuos convalecientes o vacunados con la vacuna de Pfizer (ver estudio).
La preocupación con esta variante en Brasil llevó a incluso recomendar a personas jóvenes acudir al primer día de síntomas al Hospital, por si pudieran necesitar intubación (ver noticia).
VARIANTE B.1.427/B.1.429/CAL.20C APARECIDA EN CALIFORNIA, ESTADOS UNIDOS
En julio de 2020 se detectó una variante en California definida por cinco mutaciones, una de ellas en la proteína ORF1a (I4205V), otra en la ORF1b (D1183Y), y tres en la proteína S (S13I, W152C y L452R). Sin embargo, fue en diciembre cuando se empezó a advertir su predominancia en la zona, coincidiendo con el notable aumento de casos en Los Ángeles. Además, tiene otras mutaciones en la proteína S similares a la variante británica. La mutación L452R se ha encontrado que hace a la proteína S notoriamente más resistente a los anticuerpos monoclonales, y podría ser resistente al suero de pacientes convalecientes o de personas vacunadas (ver estudio). Esta variante se detectó en España en marzo de 2021 (ver noticia).
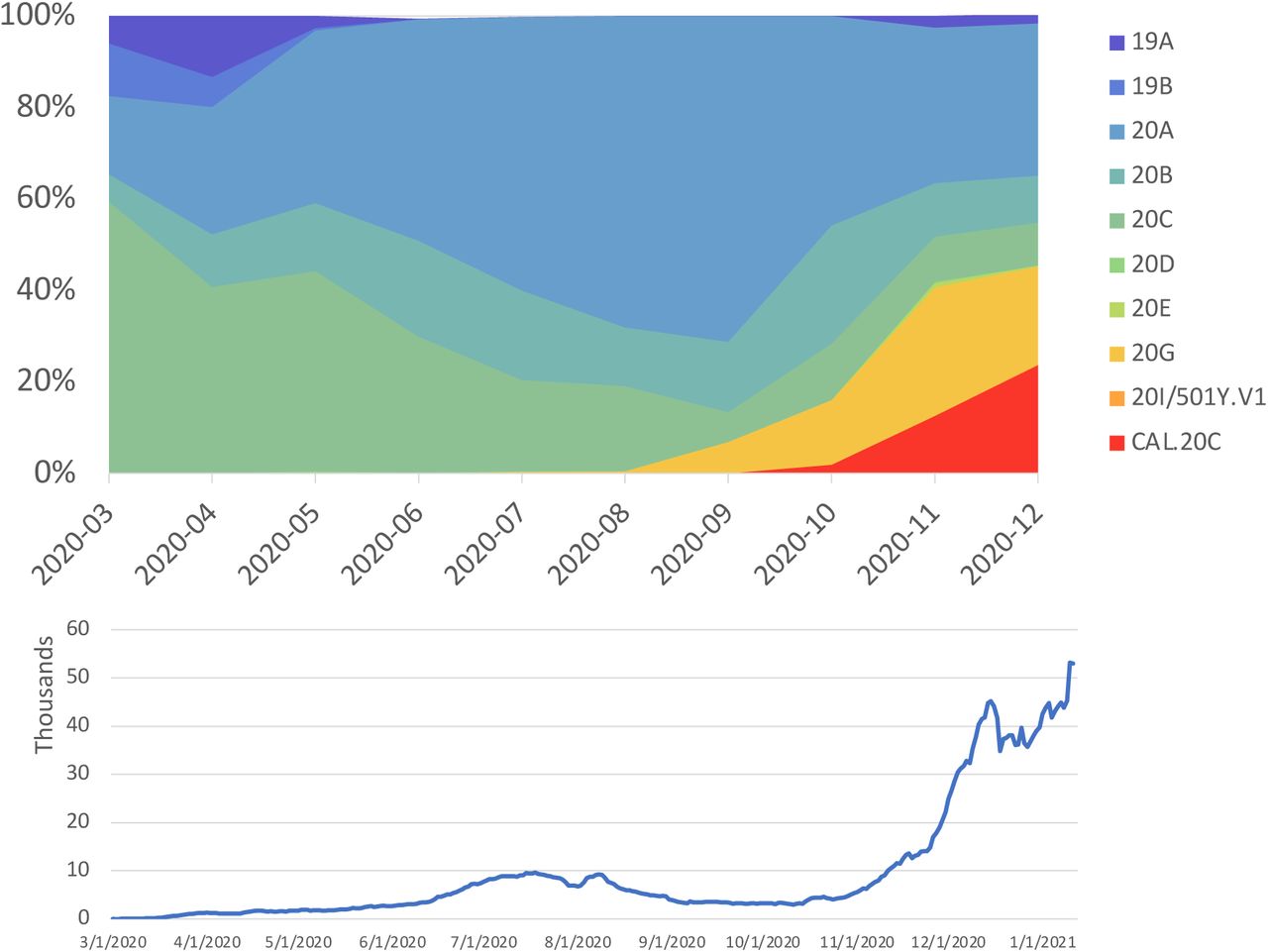
Evolución de la variante CAL20C en California. Fuente: medrxiv
VARIANTE A.23.1 EN UGANDA
En febrero de 2021 se detectó una variante del coronavirus en Uganda, con mutaciones similar a la variante británica. Esta variante evolucionó de manera independiente a la variante del Reino Unido, que es del "linaje B", mientras que la de Uganda es del "linaje A". Según el GISAID, tiene una serie de variantes de "potencial preocupación biológica", y se había hecho predominante en Kampala. Se ha identificado una determinada mutación, P681R, que podría significar que se comporta de forma similar a la variante del Reino Unido, B.1.1.7, que tiene una mutación similar en su proteína de punta, la P681H. Las variantes A.23.1 se han detectado en otros países además de Uganda, como Reino Unido, Ruanda, Canadá y Camboya.
VARIANTE B.1.526 (NUEVA YORK)
En febrero de 2021 se identificó una nueva variante en Nueva York, con cambios en sitios críticos de unión a anticuerpos. Otras mutaciones detectadas en la proteína S son L5F, T95I, D253G, E484K o S477N, D614G y A701V (ver estudio).
VARIANTE B.1.525 - NIGERIA
Las vacunas podrían ser menos efectivas contra esta variante. Un estudio británico la coloca como LA VARIANTE MÁS MORTÍFERA CON UNA LETALIDAD DEL 4,5%.
VARIANTE B.1.617 O DELTA- INDIA
En abril, la situación en India empeoró notablemente. La variante B.1.617 parecía ser responsable de más del 70% de los nuevos casos de COVID19 en este país, llegando a registrar cientos de miles de infecciones diarias pese al avance en la vacunación, y teniendo que ser incinerados cadáveres incluso en la vía pública ante el desbordamiento de los crematorios. Esta variante ya se había detectado en Europa en abril (ver artículo).
En Mayo, tras un vertiginoso ascenso del número de casos, comenzó a descender la curva de contagios en India.
Con respecto a la eficacia de las vacunas, según datos de Reino Unido, una única dosis de Pfizer o Astra Zeneca solo sería un 33% eficaz contra esta variante (con la salvedad de que esta eficacia sería más prolongada en el tiempo en el caso de Astra Zeneca al estar mucho más separadas en el tiempo ambas dosis), y siendo la doble dosis de Pfizer más eficaz frente a esta variante (88%) que una doble dosis de Astra (60%) (ver estudio).
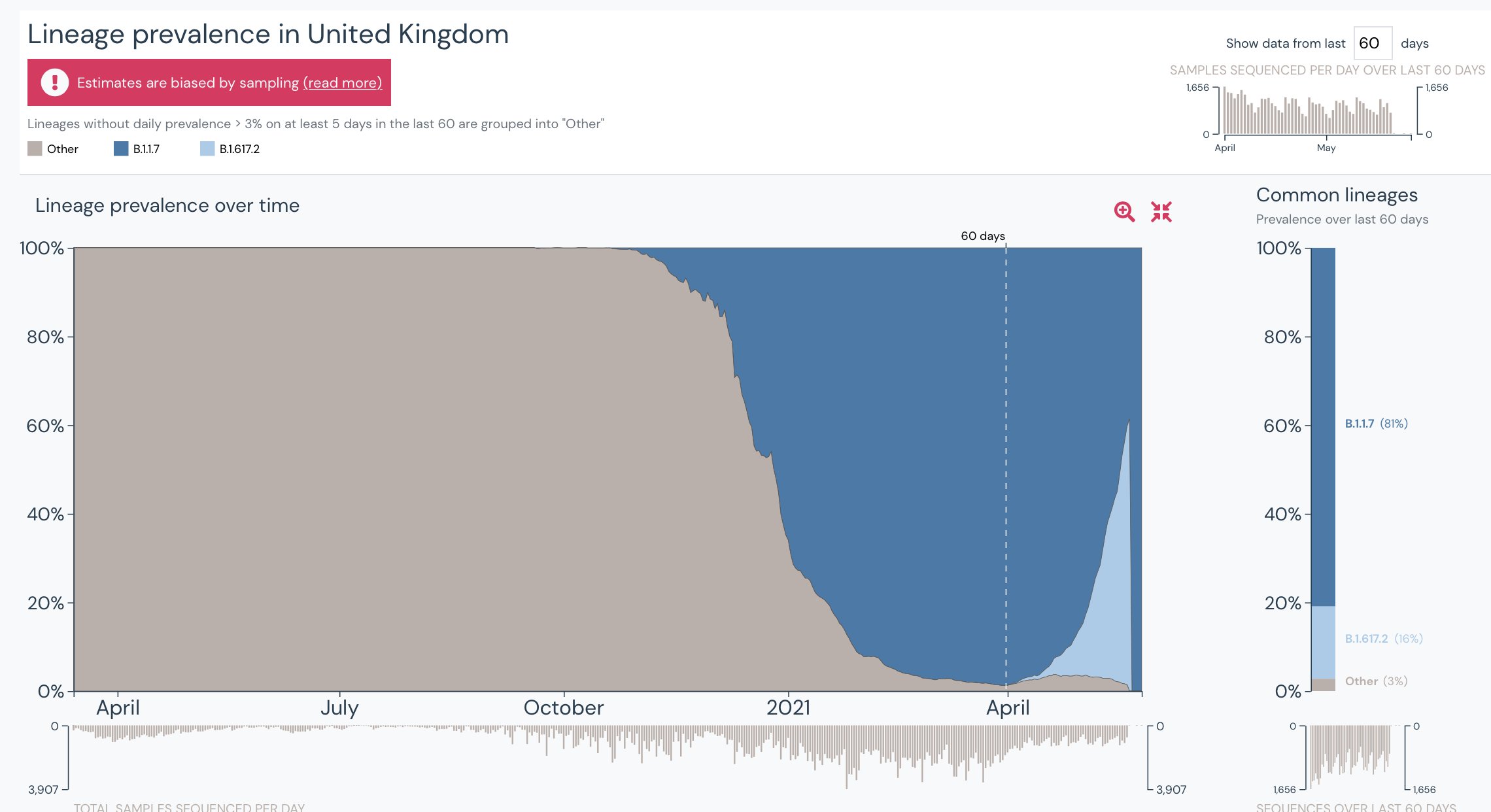
En Reino Unido, se observó que la variante de Kent, más transmisible, se convirtió en variante dominante. La agresiva campaña de vacunación agresiva bloqueó en gran medida su impacto. Posteriormente, en mayo de 2021, con un 59% de vacunados (con primera dosis), la variante B.1.617.2 estaba en aumento, superando a la variante británica.
Esta variante consiguió cambiar la tendencia del virus en muchos países. En junio de 2021, esta variante se hizo dominante en Reino Unido, y durante el verano, se hizo dominante en otros países como España, Holanda, Portugal, etc. Provocó notables subidas de contagios en todos estos países, afectando incluso a personas vacunadas con pauta completa, muchos de ellos sanitarios. Muchos de estos vacunados positivos, en la mayoría de casos no enfermaban gravemente, aunque no hay que negar que se dieron algunas hospitalizaciones, incluso en UCIs. Pero lo preocupante es que podrían estar transmitiendo el virus a personas de su entorno no vacunadas, las cuales podían enfermar más.
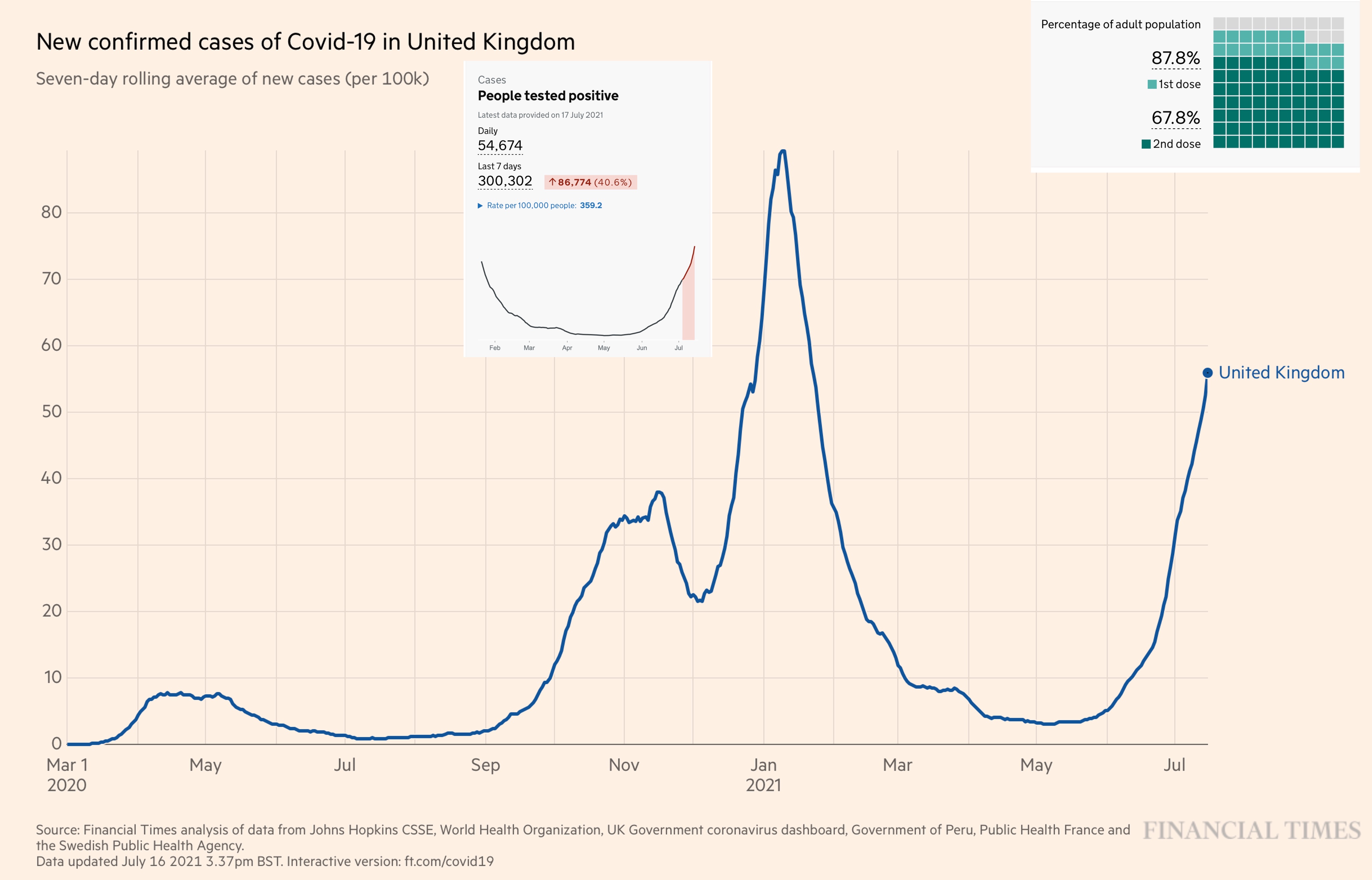
Gráfico de casos en Reino Unido (julio 2021).
Pese a contar con más del 50% con pauta completa, en julio, la extensión de la variante delta provocaba una brutal subida de casos.
Estudios señalaban que la variante delta es menos sensible a los anticuerpos neutralizantes en sueros de individuos recuperados (in vitro, 8 veces menos que la variante original), y mostraba una mayor eficacia de replicación en comparación con la variante Alfa, y cargas virales más altas en vías respiratorias que otras variantes previas. Además, los anticuerpos con capacidad de neutralizar la variante delta del SARS-CoV-2 fueron significativamente más bajos en los participantes vacunados con Astra Zeneca (ChadOx-1), en comparación con Pfizer (BNT162b2). (ver estudio)
VARIANTE C.37 - CHILE Y PERÚ
Detectada en estos países sudamericanos (ver artículo). En diciembre 2020 se observó en Lima una variante descendiente de B.1.1.1 que acumula varias mutaciones nuevas en su genoma, incluyendo algunas de las variantes de preocupación (VOCs) B.1.1.7, B.1.351 y P.1, y observándose un rápido crecimiento en la capital peruana. Las variantes C.37 y P.1 comparten la mutación ORF1a:3675-3677 y (en principio) darían el mismo resultado en una prueba de PCR, por lo que se especulaba con la posibilidad de que muchas de las muestras identificadas como P.1 fueran realmente C.37. La aparición y expansión de este linaje parecían cuadrar con el aumento de casos en varias regiones.
MUTACIONES RECURRENTES
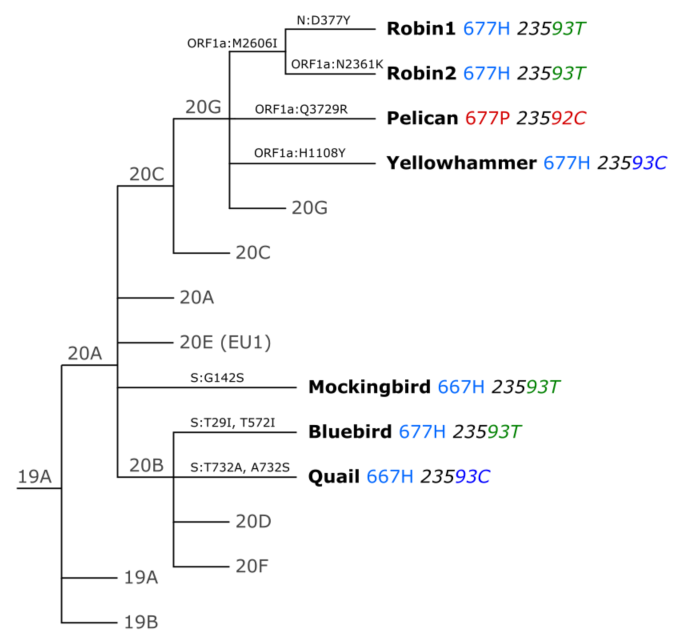
En Estados Unidos se encontró que el virus había mutado en la posición 677 de la proteína espiga en al menos siete grupos distintos, en los últimos meses (ver estudio). Esto explicaría cómo funciona el virus. Se detectó de forma independiente la mutación Q677P en Nuevo México y Luisiana, y al investigar, se descubrió un aumento de las mutaciones Q677H y Q677P en todo el mundo y dentro de los EE. UU., desde el otoño de 2020. Se hizo evidente que había múltiples ramas separadas donde las mutaciones Q677 habían surgido de forma independiente. En el estudio enlazado, centrado en los de EE. UU., se detectaron seis cambios a 677H, pero cuatro de ellos lo hacen a través del cambio a Timina en el nucleótido 23593 del ARN del virus, dos lo hacen con un cambio a Citosina en la misma posición, y otra variante cambia a 677P con un cambio a citosina en el nucleótido 23592. A estas variantes se le pusieron nombres de aves americanas.
La siguiente figura muestra todas las mutaciones de esas variantes, y las variantes Robins, Pelican y Yellowhammer provienen del linaje 20G, por lo que comparten muchas mutaciones en virtud de un ancestro común.
El hecho de que la mutación Q677 esté apareciendo de forma independiente varias veces podría significar un ejemplo de evolución convergente o paralela, lo que sugiere que la mutación puede ofrecer algún tipo de beneficio al virus, de modo que cuando aparece (las mutaciones aparecen de manera bastante aleatoria) se selecciona positivamente, por lo que su frecuencia aumenta.
También se estaba encontrando que la aparición de la mutación Nelly (N501Y) y Erik (E484K) estaban siendo recurrentes.
Fuente: The economist
Si bien estas mutaciones no presentan una dinámica como las variantes de interés (VoC) antes comentadas (501Y.V1 (B.1.17) o 501Y.V2), y tampoco está claro que deba haber algún impacto en la inmunidad o cambio en el resultado clínico, podría ser un claro ejemplo de que el virus también continúa adaptándose, por lo que la vigilancia de las mutaciones es importante para saber qué entornos y fuerzas conducen a estos cambios. ¿Adaptación general a los humanos? ¿Respuestas al aumento de la inmunidad? ¿Ganancias directas en la transmisión?.
A continuación puedes ver varias imágenes que resumen las principales características de las variantes de preocupación.

Fuente: @erictopol
Mutaciones significativas de las variantes británica, sudafricana y brasileña
A primeros de febrero de 2021, se especulaba también con la posibilidad de aparición de otra variante mexicana, que incluía la mutación E484K (ver noticia).
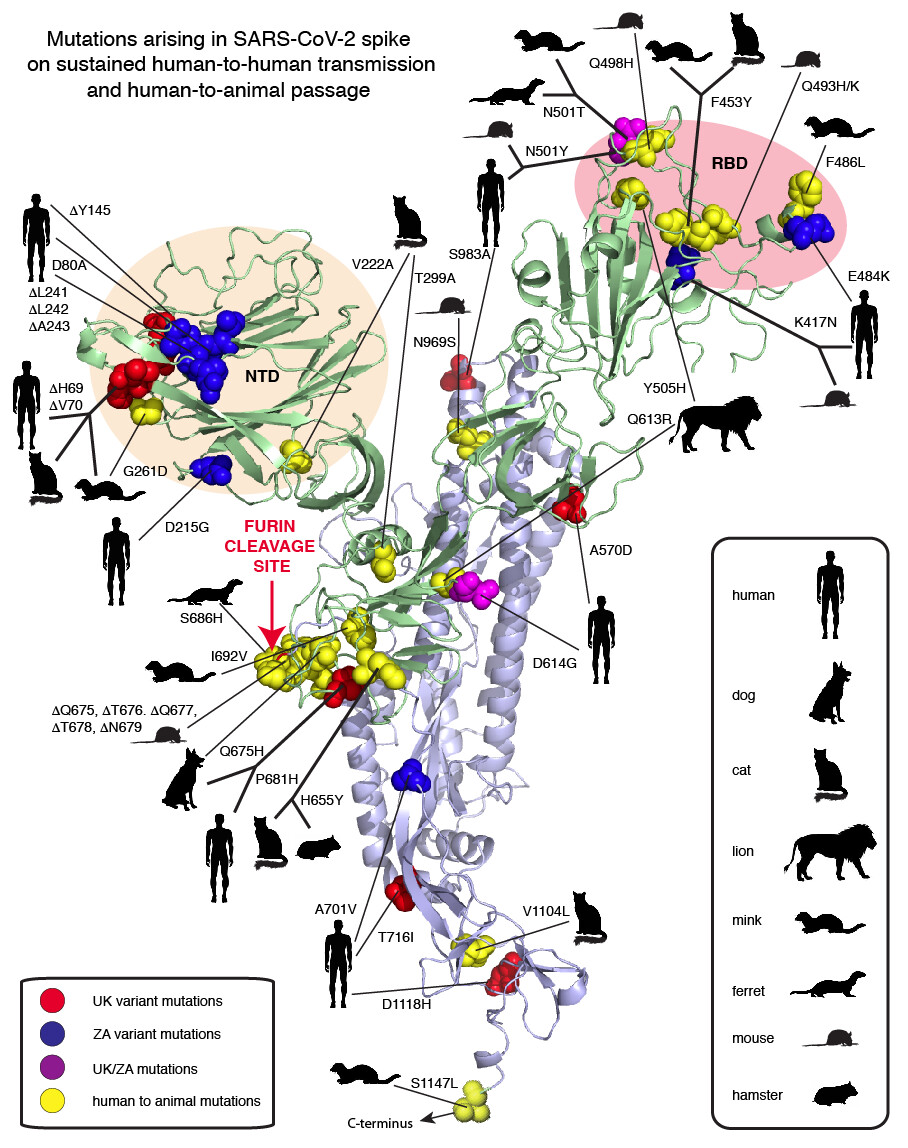
En enero de 2021, en la web virological.org, se recogieron en un artículo las mutaciones de la proteína S más representativas, surgidas en la transmisión paso de persona a persona y de persona a animal, que incluyen (ver artículo):
hCoV-19 / SouthAfrica / Tygerberg-461/2020 | EPI_ISL_745186 | 2020-12-07
hCoV-19 / England / LOND- 1267020/2020 | EPI_ISL_741243 | 2020-12-11
hCoV-19 / mouse / Harbin / HRB-26m / 2020 | EPI_ISL_459910 | 2020-04-19
hCoV-19 / mink / Países Bajos / 1/2020 | EPI_ISL_431778 | 2020-04- 24
hCoV-19 / mink / Países Bajos / NB01_02KS / 2020 | EPI_ISL_447624 | 2020-04-29
hCoV-19 / mink / Países Bajos / NB02_07KS / 2020 | EPI_ISL_447629 | 2020-04-29
hCoV-19 / mink / Países Bajos / NB02_16RS / 2020 | EPI_ISL_447632 | 2020-04-28
hCoV-19 / cat / France / Env-Ba / 2020 | EPI_ISL_483063 | 2020-05-14
hCoV-19 / cat / France / Env-Di / 2020 | EPI_ISL_483064 | 2020-05-14
hCoV-19 / cat / Bélgica / BE-MG-0320/2020 | EPI_ISL_487275 | 2020-03-11
hCoV-19 / cat / Dinamarca / mDK-315/2020 | EPI_ISL_683164 | 2020-11-17
hCoV-19 / cat / USA / TX-TAMU-013/2020 | EPI_ISL_699506 | 2020-06-28
hCoV-19 / cat / USA / TX-TAMU-057/2020 | EPI_ISL_699507 | 2020-07-17
hCoV-19 / cat / USA / TX- TAMU-078/2020 | EPI_ISL_699509 | 2020-07-29
hCoV-19 / cat / Greece / 2K / 2020 | EPI_ISL_717979 | 2020-11-23
hCoV-19 / lion / USA / NY-3-041520 / 2020 | EPI_ISL_566037 | 2020-04-04
hCoV-19 / lion / USA / NY-041520/2020 | EPI_ISL_566038 | 2020-04-04
hCoV-19 / lion / USA / NY-2/2020 | EPI_ISL_566044 | 2020-04-04
hCoV-19 / tiger / USA / NY-040420/2020 | EPI_ISL_420293 | 2020-04-02
hCoV-19 / dog / HongKong / 20-02756 / 2020 | EPI_ISL_414518 | 2020-02-26
hCoV-19 / dog / USA / TX-TAMU -077/2020 | EPI_ISL_699508 | 2020-07-28
hCoV-19 / dog / Italia / Perro399-20BA / 2020 | EPI_ISL_730652 | 2020-11-04,
En mayo de 2021, se publicó un estudio en el que se proponía que una mutación, la T403R de la proteína S, permitía al coronavirus de murciélago RaTG13 unirse al receptor ACE2 humano, pudiendo ser el requisito necesario previo a una transmisión zoonótica del SARS-CoV-2 (ver estudio).
















 Factores de riesgo
Factores de riesgo Técnicas de detección
Técnicas de detección Tratamientos
Tratamientos Vacunas
Vacunas Carga viral
Carga viral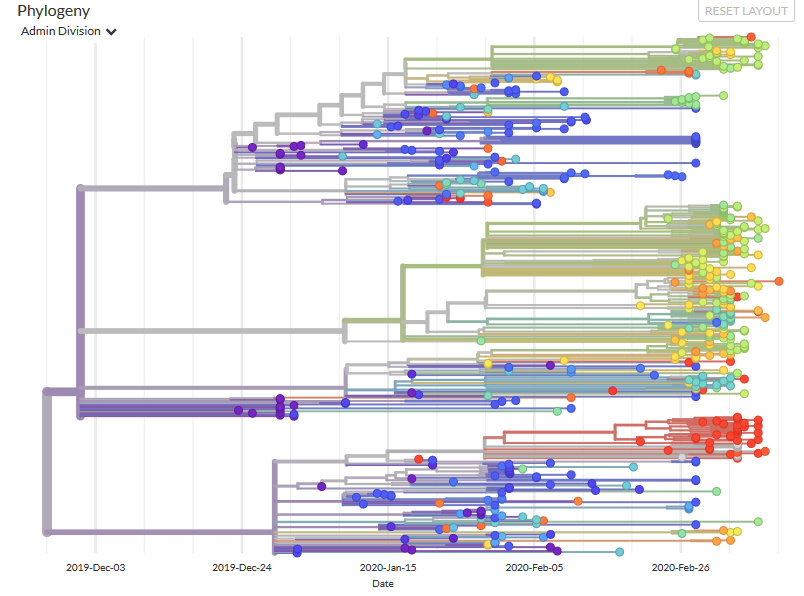 Mutaciones
Mutaciones Letalidad y mortalidad
Letalidad y mortalidad Prevención: mascarillas
Prevención: mascarillas Prevención: Ventilación y filtración (HEPA)
Prevención: Ventilación y filtración (HEPA) Otras medidas de prevención
Otras medidas de prevención Impacto de la pandemia
Impacto de la pandemia Fin de la pandemia
Fin de la pandemia Prevención en centros educativos
Prevención en centros educativos Enlaces de interés
Enlaces de interés





